Mol:FL5FACGL0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2929 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2929 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2929 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2929 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5919 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5919 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8909 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8909 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8909 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8909 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5919 1.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5919 1.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1899 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1899 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4889 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4889 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4889 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4889 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1899 1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1899 1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1899 -0.7024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1899 -0.7024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2119 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2119 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9264 1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9264 1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6409 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6409 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6409 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6409 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9264 2.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9264 2.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2119 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2119 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9937 1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9937 1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0711 -0.2817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0711 -0.2817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5919 -0.8804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5919 -0.8804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4397 2.8337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4397 2.8337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4382 -1.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4382 -1.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2962 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2962 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0632 -1.7502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0632 -1.7502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2962 -2.5706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2962 -2.5706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4382 -2.9947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4382 -2.9947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2052 -2.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2052 -2.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4146 -0.7101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4146 -0.7101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6850 -2.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6850 -2.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3059 -3.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3059 -3.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2466 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2466 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8041 -1.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8041 -1.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6068 -0.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6068 -0.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3815 -0.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3815 -0.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8186 -0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8186 -0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1162 -0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1162 -0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1838 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1838 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1645 -1.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1645 -1.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9937 -0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9937 -0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9264 3.6097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9264 3.6097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3073 -3.6097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3073 -3.6097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5671 -3.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5671 -3.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.0111 1.0392 | + | M SBV 1 46 0.0111 1.0392 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0005 | + | ID FL5FACGL0005 |
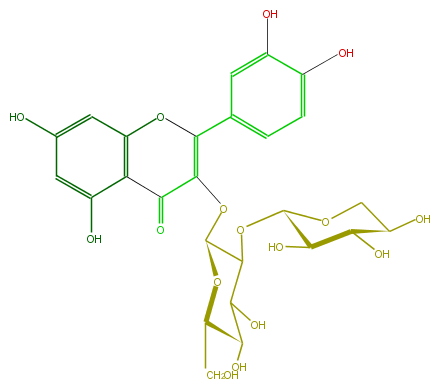
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O | + | SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.2929 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2929 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5919 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8909 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8909 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5919 1.5476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1899 -0.0713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4889 0.3335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4889 1.1429 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1899 1.5476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1899 -0.7024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2119 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9264 1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6409 1.5475 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6409 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9264 2.7850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2119 2.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9937 1.5475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0711 -0.2817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5919 -0.8804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4397 2.8337 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4382 -1.3589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2962 -0.9349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0632 -1.7502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2962 -2.5706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4382 -2.9947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2052 -2.1793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4146 -0.7101 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6850 -2.6195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3059 -3.4405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2466 -0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8041 -1.0726 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6068 -0.7604 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3815 -0.7521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8186 -0.1890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1162 -0.5598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1838 -1.0545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1645 -1.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9937 -0.4862 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9264 3.6097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3073 -3.6097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5671 -3.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.0111 1.0392
S SKP 5
ID FL5FACGL0005
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O
M END