Mol:BMCCCC--a001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 63 67 0 0 1 0 0 0 0 0999 V2000 | + | 63 67 0 0 1 0 0 0 0 0999 V2000 |
| − | 3.6092 -9.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6092 -9.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7431 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7431 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7431 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7431 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6092 -11.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6092 -11.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4752 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4752 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4752 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4752 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6092 -8.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6092 -8.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4752 -8.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4752 -8.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4752 -7.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4752 -7.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3412 -6.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3412 -6.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.3412 -5.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.3412 -5.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -11.8567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -11.8567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4067 -12.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4067 -12.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4013 -12.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4013 -12.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4752 -5.1876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4752 -5.1876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.0456 11.0761 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.0456 11.0761 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.7365 10.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.7365 10.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.7584 9.9171 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.7584 9.9171 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.0893 10.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.0893 10.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.3983 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.3983 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.3764 11.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.3764 11.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.5893 12.1991 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.5893 12.1991 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.7802 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.7802 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.0893 10.6603 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.0893 10.6603 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.6854 12.7703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.6854 12.7703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 14.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 13.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 13.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 13.1925 8.9002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 13.1925 8.9002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 14.0015 8.3124 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 14.0015 8.3124 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 14.0015 7.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.0015 7.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.9137 10.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.9137 10.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2414 8.5912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2414 8.5912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8105 8.9002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8105 8.9002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1354 6.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1354 6.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4983 9.2603 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4983 9.2603 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.8291 8.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.8291 8.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1674 10.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1674 10.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.7551 9.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.7551 9.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1354 5.8124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1354 5.8124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.1354 4.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.1354 4.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2694 4.3124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2694 4.3124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.7694 5.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.7694 5.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.7694 3.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.7694 3.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4034 3.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4034 3.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.4034 2.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.4034 2.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5374 2.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5374 2.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.0374 3.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.0374 3.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.0374 1.4464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.0374 1.4464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.6713 1.8124 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 9.6713 1.8124 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 9.6713 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.6713 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8053 0.3124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8053 0.3124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8053 -0.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8053 -0.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9393 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9393 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9393 -2.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9393 -2.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0733 -2.6876 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0733 -2.6876 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0733 -3.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0733 -3.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2072 -4.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2072 -4.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8053 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8053 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.5374 0.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.5374 0.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.8053 -2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.8053 -2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2072 -5.1876 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2072 -5.1876 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16 21 2 0 0 0 0 | + | 16 21 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 20 19 2 0 0 0 0 | + | 20 19 2 0 0 0 0 |
| − | 24 23 1 0 0 0 0 | + | 24 23 1 0 0 0 0 |
| − | 23 22 2 0 0 0 0 | + | 23 22 2 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 21 25 1 0 0 0 0 | + | 21 25 1 0 0 0 0 |
| − | 29 33 1 6 0 0 0 | + | 29 33 1 6 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 26 33 1 6 0 0 0 | + | 26 33 1 6 0 0 0 |
| − | 28 27 1 0 0 0 0 | + | 28 27 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 26 24 1 0 0 0 0 | + | 26 24 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 39 41 2 0 0 0 0 | + | 39 41 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 46 43 1 0 0 0 0 | + | 46 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 43 42 1 0 0 0 0 | + | 43 42 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 51 60 1 6 0 0 0 | + | 51 60 1 6 0 0 0 |
| − | 52 61 2 0 0 0 0 | + | 52 61 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 56 62 2 0 0 0 0 | + | 56 62 2 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | 27 31 1 1 0 0 0 | + | 27 31 1 1 0 0 0 |
| − | 28 32 1 1 0 0 0 | + | 28 32 1 1 0 0 0 |
| − | 37 35 1 0 0 0 0 | + | 37 35 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 32 35 1 0 0 0 0 | + | 32 35 1 0 0 0 0 |
| − | 13 12 1 0 0 0 0 | + | 13 12 1 0 0 0 0 |
| − | 4 14 1 0 0 0 0 | + | 4 14 1 0 0 0 0 |
| − | 14 13 1 0 0 0 0 | + | 14 13 1 0 0 0 0 |
| − | 12 3 1 0 0 0 0 | + | 12 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 3 2 1 0 0 0 0 | + | 3 2 1 0 0 0 0 |
| − | 2 1 2 0 0 0 0 | + | 2 1 2 0 0 0 0 |
| − | 1 6 1 0 0 0 0 | + | 1 6 1 0 0 0 0 |
| − | 6 5 2 0 0 0 0 | + | 6 5 2 0 0 0 0 |
| − | 5 4 1 0 0 0 0 | + | 5 4 1 0 0 0 0 |
| − | 1 7 1 0 0 0 0 | + | 1 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 15 2 0 0 0 0 | + | 11 15 2 0 0 0 0 |
| − | 11 63 1 0 0 0 0 | + | 11 63 1 0 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMCCCC--a001 | + | ID BMCCCC--a001 |
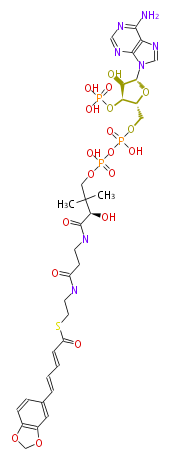
| − | NAME (E,E)-Piperoyl-CoA | + | NAME (E,E)-Piperoyl-CoA |
| − | FORMULA C33H44N7O19P3S | + | FORMULA C33H44N7O19P3S |
| − | EXACTMASS 967.1625 | + | EXACTMASS 967.1625 |
| − | AVERAGEMASS 967.7255 | + | AVERAGEMASS 967.7255 |
| − | SMILES c(O5)(c4OC5)cc(cc4)C=CC=CC(SCCNC(=O)CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@H]([C@@H]1n(c23)cnc2c(ncn3)N)O)(O)=O)O)=O)=O | + | SMILES c(O5)(c4OC5)cc(cc4)C=CC=CC(SCCNC(=O)CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@H]([C@@H]1n(c23)cnc2c(ncn3)N)O)(O)=O)O)=O)=O |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02611 | + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02611 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
63 67 0 0 1 0 0 0 0 0999 V2000
3.6092 -9.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7431 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7431 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6092 -11.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4752 -11.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4752 -10.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6092 -8.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4752 -8.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4752 -7.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3412 -6.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.3412 -5.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -11.8567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4067 -12.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4013 -12.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4752 -5.1876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.0456 11.0761 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
17.7365 10.1250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.7584 9.9171 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
16.0893 10.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
16.3983 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
17.3764 11.8192 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.5893 12.1991 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
14.7802 11.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
15.0893 10.6603 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
17.6854 12.7703 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
14.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
13.5015 9.8512 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
13.1925 8.9002 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
14.0015 8.3124 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
14.0015 7.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
12.9137 10.6603 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.2414 8.5912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.8105 8.9002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.1354 6.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.4983 9.2603 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
10.8291 8.5171 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.1674 10.0034 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.7551 9.9294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.1354 5.8124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
12.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
14.1354 5.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.1354 4.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.2694 4.3124 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
11.7694 5.1784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.7694 3.4464 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.4034 3.8124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
11.4034 2.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.5374 2.3124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.0374 3.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.0374 1.4464 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.6713 1.8124 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
9.6713 0.8124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8053 0.3124 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
8.8053 -0.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9393 -1.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.9393 -2.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0733 -2.6876 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.0733 -3.6876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2072 -4.1876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.8053 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
10.5374 0.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.8053 -2.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.2072 -5.1876 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
16 21 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 19 1 0 0 0 0
20 21 1 0 0 0 0
20 19 2 0 0 0 0
24 23 1 0 0 0 0
23 22 2 0 0 0 0
22 20 1 0 0 0 0
19 24 1 0 0 0 0
21 25 1 0 0 0 0
29 33 1 6 0 0 0
28 29 1 0 0 0 0
26 33 1 6 0 0 0
28 27 1 0 0 0 0
26 27 1 0 0 0 0
29 30 1 0 0 0 0
30 34 1 0 0 0 0
26 24 1 0 0 0 0
34 39 1 0 0 0 0
39 40 1 0 0 0 0
39 41 2 0 0 0 0
39 42 1 0 0 0 0
46 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
43 42 1 0 0 0 0
47 48 1 0 0 0 0
48 51 1 0 0 0 0
48 49 1 0 0 0 0
48 50 1 0 0 0 0
51 52 1 0 0 0 0
51 60 1 6 0 0 0
52 61 2 0 0 0 0
52 53 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
56 62 2 0 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 63 1 0 0 0 0
27 31 1 1 0 0 0
28 32 1 1 0 0 0
37 35 1 0 0 0 0
35 38 1 0 0 0 0
35 36 2 0 0 0 0
32 35 1 0 0 0 0
13 12 1 0 0 0 0
4 14 1 0 0 0 0
14 13 1 0 0 0 0
12 3 1 0 0 0 0
3 4 2 0 0 0 0
3 2 1 0 0 0 0
2 1 2 0 0 0 0
1 6 1 0 0 0 0
6 5 2 0 0 0 0
5 4 1 0 0 0 0
1 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 15 2 0 0 0 0
11 63 1 0 0 0 0
S SKP 7
ID BMCCCC--a001
NAME (E,E)-Piperoyl-CoA
FORMULA C33H44N7O19P3S
EXACTMASS 967.1625
AVERAGEMASS 967.7255
SMILES c(O5)(c4OC5)cc(cc4)C=CC=CC(SCCNC(=O)CCNC([C@@H](C(C)(C)COP(OP(O)(=O)OC[C@@H](O1)[C@@H](OP(O)(O)=O)[C@H]([C@@H]1n(c23)cnc2c(ncn3)N)O)(O)=O)O)=O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C02611
M END