Mol:BMCCPPHM0003
From Metabolomics.JP
(Difference between revisions)
| (4 intermediate revisions by one user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | + | 59 66 0 0 0 0 0 0 0 0999 V2000 | |
| − | + | 1.5207 -0.2571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.3387 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.7499 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.5680 -0.2571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.0828 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.2571 0.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.8170 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.8170 1.3387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.2571 1.5207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.0828 2.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.5680 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.7499 2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.3387 2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.5207 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.1714 2.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.3457 1.5207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.9057 1.3387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.9057 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.3457 0.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.1714 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.8237 -1.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.2650 -1.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.6006 -2.2382 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.1156 -2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.4512 -3.6594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.0337 -4.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.3018 -5.0807 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.1832 -5.7481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.5556 -1.3983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.2717 -3.7457 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -1.4845 0.2650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -1.4845 1.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -2.2383 1.4881 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -2.9058 1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -3.6594 1.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -4.3269 2.1224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -5.0807 1.7869 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -5.7481 2.2718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -3.7457 0.8170 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.2650 3.5732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.8237 3.5732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.4881 4.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.9730 4.9944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 3.5732 1.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 4.3269 1.4881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 4.9944 1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 3.5732 0.2650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.0444 0.2193 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.8694 1.0444 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.0444 1.8694 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 0.2193 1.0444 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.0444 1.0444 0.0000 Fe 0 1 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -4.2407 2.9430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -0.8543 -4.2407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 4.9082 2.7936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 5.7481 1.6375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 2.7935 4.9082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 1.6375 5.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | -1.4845 2.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | |
| − | + | 3 4 2 0 | |
| − | + | 3 2 1 0 | |
| − | + | 2 1 2 0 | |
| − | 4 48 1 | + | 1 48 1 0 |
| − | 1 20 1 | + | 4 48 1 0 |
| − | 20 19 2 | + | 1 20 1 0 |
| − | 19 18 1 | + | 20 19 2 0 |
| − | 18 17 2 | + | 19 18 1 0 |
| − | 19 49 1 | + | 18 17 2 0 |
| − | 49 16 2 | + | 19 49 1 0 |
| − | 16 17 1 | + | 49 16 2 0 |
| − | 16 15 1 | + | 16 17 1 0 |
| − | 15 14 2 | + | 16 15 1 0 |
| − | 14 13 1 | + | 15 14 2 0 |
| − | 14 50 1 | + | 14 13 1 0 |
| − | 50 11 1 | + | 14 50 1 0 |
| − | 11 12 1 | + | 50 11 1 0 |
| − | 12 13 2 | + | 11 12 1 0 |
| − | 11 10 2 | + | 12 13 2 0 |
| − | 10 9 1 | + | 11 10 2 0 |
| − | 9 51 2 | + | 10 9 1 0 |
| − | 51 6 1 | + | 9 51 2 0 |
| − | 6 7 1 | + | 51 6 1 0 |
| − | 7 8 2 | + | 6 7 1 0 |
| − | 8 9 1 | + | 7 8 2 0 |
| − | 4 5 1 | + | 8 9 1 0 |
| − | 5 6 2 | + | 4 5 1 0 |
| − | 48 52 1 | + | 5 6 2 0 |
| − | 52 51 1 | + | 48 52 1 0 |
| − | 52 49 1 | + | 52 51 1 0 |
| − | 52 50 1 | + | 52 49 1 0 |
| − | 7 31 1 | + | 52 50 1 0 |
| − | 8 32 1 | + | 7 31 1 0 |
| − | 32 33 1 | + | 8 32 1 0 |
| − | 33 34 1 | + | 32 33 1 0 |
| − | 34 35 1 | + | 33 34 1 0 |
| − | 35 36 1 | + | 34 35 1 0 |
| − | 36 37 1 | + | 35 36 1 0 |
| − | 36 53 2 | + | 36 37 1 0 |
| − | 35 39 1 4 | + | 36 53 2 0 |
| − | 37 38 1 | + | 35 39 1 4 |
| − | 3 22 1 | + | 37 38 1 0 |
| − | 22 29 1 4 | + | 3 22 1 0 |
| − | 22 23 1 | + | 22 29 1 4 |
| − | 23 24 1 | + | 22 23 1 0 |
| − | 24 25 1 | + | 23 24 1 0 |
| − | 25 26 1 | + | 24 25 1 0 |
| − | 25 30 1 4 | + | 25 26 1 0 |
| − | 26 27 1 | + | 25 30 1 4 |
| − | 26 54 2 | + | 26 27 1 0 |
| − | 27 28 1 | + | 26 54 2 0 |
| − | 2 21 1 | + | 27 28 1 0 |
| − | 18 47 1 | + | 2 21 1 0 |
| − | 17 44 1 | + | 18 47 1 0 |
| − | 44 45 1 | + | 17 44 1 0 |
| − | 45 46 1 | + | 44 45 1 0 |
| − | 46 55 1 | + | 45 46 1 0 |
| − | 46 56 2 | + | 46 55 1 0 |
| − | 13 41 1 | + | 46 56 2 0 |
| − | 41 42 1 | + | 13 41 1 0 |
| − | 42 43 1 | + | 41 42 1 0 |
| − | 43 57 2 | + | 42 43 1 0 |
| − | 43 58 1 0 | + | 43 57 2 0 |
| − | + | 43 58 1 0 | |
| − | S SKP | + | 12 40 1 0 |
| − | ID BMCCPPHM0003 | + | 32 59 1 4 |
| − | NAME | + | M CHG 1 52 3 |
| − | FORMULA | + | S SKP 6 |
| − | EXACTMASS | + | ID BMCCPPHM0003 |
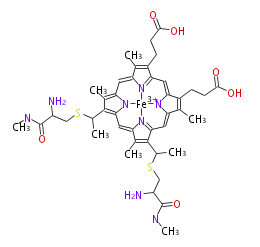
| − | AVERAGEMASS | + | NAME 3- [7,12-bis[1- [2-amino-3- (methylamino) -3-oxopropyl] sulfanylethyl] -18- (2-carboxyethyl) -3,8,13,17-tetramethylporphyrin-21,23-diid-2-yl] propanoic acid; iron(3+) |
| − | SMILES | + | FORMULA C42H52FeN8O6S2 |
| − | + | EXACTMASS 884.2800649159999 | |
| + | AVERAGEMASS 884.8896 | ||
| + | SMILES C(C(C)SCC(N)C(=O)NC)(=C1C)C(C=4)=N([Fe+3]623)C1=Cc(c(C(C)SCC(N)C(NC)=O)8)n2c(c8C)C=C(C(C)=7)N3=C(C7CCC(O)=O)C=c(n65)c(c(c45)C)CCC(O)=O | ||
M END | M END | ||
| − | |||
Latest revision as of 11:42, 16 June 2010
Copyright: ARM project http://www.metabolome.jp/
59 66 0 0 0 0 0 0 0 0999 V2000
1.5207 -0.2571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3387 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7499 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5680 -0.2571 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0828 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2571 0.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8170 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8170 1.3387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2571 1.5207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0828 2.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5680 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7499 2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3387 2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5207 2.3457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1714 2.1714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3457 1.5207 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9057 1.3387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9057 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3457 0.5680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1714 -0.0827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8237 -1.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2650 -1.4845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6006 -2.2382 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
0.1156 -2.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4512 -3.6594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0337 -4.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3018 -5.0807 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-0.1832 -5.7481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5556 -1.3983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2717 -3.7457 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-1.4845 0.2650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4845 1.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2383 1.4881 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-2.9058 1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6594 1.6375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3269 2.1224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0807 1.7869 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
-5.7481 2.2718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7457 0.8170 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
0.2650 3.5732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8237 3.5732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4881 4.3269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9730 4.9944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5732 1.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3269 1.4881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9944 1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5732 0.2650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0444 0.2193 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
1.8694 1.0444 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
1.0444 1.8694 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
0.2193 1.0444 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
1.0444 1.0444 0.0000 Fe 0 1 0 0 0 0 0 0 0 0 0 0
-4.2407 2.9430 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8543 -4.2407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9082 2.7936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7481 1.6375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7935 4.9082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6375 5.7481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4845 2.6487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3 4 2 0
3 2 1 0
2 1 2 0
1 48 1 0
4 48 1 0
1 20 1 0
20 19 2 0
19 18 1 0
18 17 2 0
19 49 1 0
49 16 2 0
16 17 1 0
16 15 1 0
15 14 2 0
14 13 1 0
14 50 1 0
50 11 1 0
11 12 1 0
12 13 2 0
11 10 2 0
10 9 1 0
9 51 2 0
51 6 1 0
6 7 1 0
7 8 2 0
8 9 1 0
4 5 1 0
5 6 2 0
48 52 1 0
52 51 1 0
52 49 1 0
52 50 1 0
7 31 1 0
8 32 1 0
32 33 1 0
33 34 1 0
34 35 1 0
35 36 1 0
36 37 1 0
36 53 2 0
35 39 1 4
37 38 1 0
3 22 1 0
22 29 1 4
22 23 1 0
23 24 1 0
24 25 1 0
25 26 1 0
25 30 1 4
26 27 1 0
26 54 2 0
27 28 1 0
2 21 1 0
18 47 1 0
17 44 1 0
44 45 1 0
45 46 1 0
46 55 1 0
46 56 2 0
13 41 1 0
41 42 1 0
42 43 1 0
43 57 2 0
43 58 1 0
12 40 1 0
32 59 1 4
M CHG 1 52 3
S SKP 6
ID BMCCPPHM0003
NAME 3- [7,12-bis[1- [2-amino-3- (methylamino) -3-oxopropyl] sulfanylethyl] -18- (2-carboxyethyl) -3,8,13,17-tetramethylporphyrin-21,23-diid-2-yl] propanoic acid; iron(3+)
FORMULA C42H52FeN8O6S2
EXACTMASS 884.2800649159999
AVERAGEMASS 884.8896
SMILES C(C(C)SCC(N)C(=O)NC)(=C1C)C(C=4)=N([Fe+3]623)C1=Cc(c(C(C)SCC(N)C(NC)=O)8)n2c(c8C)C=C(C(C)=7)N3=C(C7CCC(O)=O)C=c(n65)c(c(c45)C)CCC(O)=O
M END