Mol:BMFYS4CAa020
From Metabolomics.JP
(Difference between revisions)
| (One intermediate revision by one user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 58 0 0 1 0 0 0 0 0999 V2000 | + | 56 58 0 0 1 0 0 0 0 0999 V2000 |
| − | 2.6691 -3.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6691 -3.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6473 -4.1024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 3.6473 -4.1024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 4.3164 -3.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3164 -3.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2946 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2946 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6517 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6517 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7856 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7856 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.7856 -1.0373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.7856 -1.0373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.6517 -0.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.6517 -0.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5177 -1.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5177 -1.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.5177 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.5177 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.2608 -0.3681 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.2608 -0.3681 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.8541 0.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.8541 0.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8596 0.4409 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8596 0.4409 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 24.3837 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 24.3837 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.1904 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 22.1904 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 22.3984 2.1622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 22.3984 2.1622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 21.5323 2.6622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 21.5323 2.6622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 20.7892 1.9930 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 20.7892 1.9930 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 19.8110 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.8110 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.8692 0.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.8692 0.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.2001 -0.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.2001 -0.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 14.9432 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 14.9432 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.4569 0.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.4569 0.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.5309 -0.8910 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 13.5309 -0.8910 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 12.5528 -0.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.5528 -0.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 11.8837 -1.4263 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 11.8837 -1.4263 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.9055 -1.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.9055 -1.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 10.2364 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 10.2364 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 9.2582 -1.7536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 9.2582 -1.7536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.5891 -2.4967 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.5891 -2.4967 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.6110 -2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.6110 -2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9418 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9418 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9637 -2.8241 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9637 -2.8241 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3601 -2.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3601 -2.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0000 -4.6377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0000 -4.6377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9563 -5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9563 -5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6036 -4.5183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6036 -4.5183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.3119 2.5689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.3119 2.5689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.4278 3.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.4278 3.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.1959 1.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.1959 1.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 19.1419 1.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 19.1419 1.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.2368 4.2445 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.2368 4.2445 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 22.8246 3.4355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 22.8246 3.4355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 21.6490 5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 21.6490 5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 23.0458 4.8323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 23.0458 4.8323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.1638 1.6657 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.1638 1.6657 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 18.3717 2.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 18.3717 2.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.9559 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.9559 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.1856 1.8736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.1856 1.8736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 16.5165 1.1305 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 | + | 16.5165 1.1305 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 17.2596 0.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 17.2596 0.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.7733 1.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.7733 1.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 15.8474 0.3873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 15.8474 0.3873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 13.8400 -1.8421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 13.8400 -1.8421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 12.2438 0.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 12.2438 0.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 8.9492 -0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 8.9492 -0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5 10 2 0 0 0 0 | + | 5 10 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 7 2 0 0 0 0 | + | 6 7 2 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 9 8 2 0 0 0 0 | + | 9 8 2 0 0 0 0 |
| − | 13 12 1 0 0 0 0 | + | 13 12 1 0 0 0 0 |
| − | 12 11 2 0 0 0 0 | + | 12 11 2 0 0 0 0 |
| − | 11 9 1 0 0 0 0 | + | 11 9 1 0 0 0 0 |
| − | 8 13 1 0 0 0 0 | + | 8 13 1 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 18 40 1 6 0 0 0 | + | 18 40 1 6 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 15 40 1 6 0 0 0 | + | 15 40 1 6 0 0 0 |
| − | 17 16 1 0 0 0 0 | + | 17 16 1 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 41 1 0 0 0 0 | + | 19 41 1 0 0 0 0 |
| − | 15 13 1 0 0 0 0 | + | 15 13 1 0 0 0 0 |
| − | 41 46 1 0 0 0 0 | + | 41 46 1 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 46 49 1 0 0 0 0 | + | 46 49 1 0 0 0 0 |
| − | 53 50 1 0 0 0 0 | + | 53 50 1 0 0 0 0 |
| − | 50 52 2 0 0 0 0 | + | 50 52 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 53 20 1 0 0 0 0 | + | 53 20 1 0 0 0 0 |
| − | 50 49 1 0 0 0 0 | + | 50 49 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 24 1 0 0 0 0 | + | 21 24 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 54 1 1 0 0 0 | + | 24 54 1 1 0 0 0 |
| − | 25 55 2 0 0 0 0 | + | 25 55 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 29 56 2 0 0 0 0 | + | 29 56 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 16 38 1 1 0 0 0 | + | 16 38 1 1 0 0 0 |
| − | 17 39 1 1 0 0 0 | + | 17 39 1 1 0 0 0 |
| − | 43 42 1 0 0 0 0 | + | 43 42 1 0 0 0 0 |
| − | 42 45 1 0 0 0 0 | + | 42 45 1 0 0 0 0 |
| − | 42 44 2 0 0 0 0 | + | 42 44 2 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 37 2 0 0 0 0 | + | 4 37 2 0 0 0 0 |
| − | 4 33 1 0 0 0 0 | + | 4 33 1 0 0 0 0 |
| − | 1 35 1 0 0 0 0 | + | 1 35 1 0 0 0 0 |
| − | 1 34 2 0 0 0 0 | + | 1 34 2 0 0 0 0 |
| − | 2 1 1 0 0 0 0 | + | 2 1 1 0 0 0 0 |
| − | 2 36 1 6 0 0 0 | + | 2 36 1 6 0 0 0 |
| − | S SKP 7 | + | S SKP 7 |
| − | ID BMFYS4CAa020 | + | ID BMFYS4CAa020 |
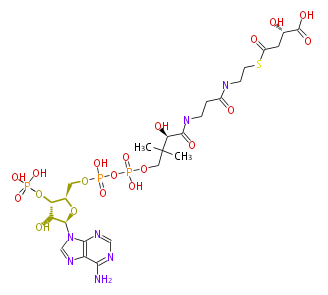
| − | NAME | + | NAME (2S) -4- [2- [3- [ [4- [ [ [(2R,3S,4R,5R) -5- (6-Aminopurin-9-yl) -4-hydroxy-3-phosphonooxyoxolan-2-yl] methoxy-hydroxyphosphoryl] oxy-hydroxyphosphoryl] oxy-2-hydroxy-3,3- dimethylbutanoyl] amino] propanoylamino] ethylsulfanyl] -2-hydroxy-4-oxobutanoic acid |
| − | FORMULA C25H40N7O20P3S | + | CAS_RN 52691-41-5 |
| − | EXACTMASS 883.1261 | + | FORMULA C25H40N7O20P3S |
| − | AVERAGEMASS 883.6075 | + | EXACTMASS 883.1261 |
| − | SMILES n(c3N)cnc(c32)n(cn2)[C@@H]([C@@H]1O)O[C@@H]([C@H]1OP(O)(O)=O)COP(OP(O)(=O)OCC([C@@H](O)C(NCCC(NCCSC(C[C@@H](C(O)=O)O)=O)=O)=O)(C)C)(O)=O | + | AVERAGEMASS 883.6075 |
| − | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04348 | + | SMILES n(c3N)cnc(c32)n(cn2)[C@@H]([C@@H]1O)O[C@@H]([C@H]1OP(O)(O)=O)COP(OP(O)(=O)OCC([C@@H](O)C(NCCC(NCCSC(C[C@@H](C(O)=O)O)=O)=O)=O)(C)C)(O)=O |
| + | KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04348 | ||
M END | M END | ||
| − | |||
Latest revision as of 10:32, 17 June 2010
Copyright: ARM project http://www.metabolome.jp/
56 58 0 0 1 0 0 0 0 0999 V2000
2.6691 -3.8945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6473 -4.1024 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
4.3164 -3.3593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2946 -3.5672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.6517 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
21.7856 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
21.7856 -1.0373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.6517 -0.5373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.5177 -1.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
23.5177 -2.0373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
24.2608 -0.3681 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
23.8541 0.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
22.8596 0.4409 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
24.3837 -2.5373 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
22.1904 1.1840 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
22.3984 2.1622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
21.5323 2.6622 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
20.7892 1.9930 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
19.8110 2.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.8692 0.5953 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.2001 -0.1479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
14.9432 -0.8170 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.4569 0.5212 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
13.5309 -0.8910 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
12.5528 -0.6831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
11.8837 -1.4263 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
10.9055 -1.2184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
10.2364 -1.9615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
9.2582 -1.7536 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
8.5891 -2.4967 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
7.6110 -2.2888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.9418 -3.0320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9637 -2.8241 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
2.3601 -2.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0000 -4.6377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9563 -5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6036 -4.5183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.3119 2.5689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.4278 3.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.1959 1.0795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
19.1419 1.4578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
22.2368 4.2445 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
22.8246 3.4355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
21.6490 5.0535 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
23.0458 4.8323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
18.1638 1.6657 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
18.3717 2.6439 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.9559 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
17.1856 1.8736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
16.5165 1.1305 0.0000 P 0 0 0 0 0 0 0 0 0 0 0 0
17.2596 0.4614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.7733 1.7996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
15.8474 0.3873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
13.8400 -1.8421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
12.2438 0.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
8.9492 -0.8025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5 10 2 0 0 0 0
5 6 1 0 0 0 0
6 7 2 0 0 0 0
7 8 1 0 0 0 0
9 10 1 0 0 0 0
9 8 2 0 0 0 0
13 12 1 0 0 0 0
12 11 2 0 0 0 0
11 9 1 0 0 0 0
8 13 1 0 0 0 0
10 14 1 0 0 0 0
18 40 1 6 0 0 0
17 18 1 0 0 0 0
15 40 1 6 0 0 0
17 16 1 0 0 0 0
15 16 1 0 0 0 0
18 19 1 0 0 0 0
19 41 1 0 0 0 0
15 13 1 0 0 0 0
41 46 1 0 0 0 0
46 48 1 0 0 0 0
46 47 2 0 0 0 0
46 49 1 0 0 0 0
53 50 1 0 0 0 0
50 52 2 0 0 0 0
50 51 1 0 0 0 0
53 20 1 0 0 0 0
50 49 1 0 0 0 0
20 21 1 0 0 0 0
21 24 1 0 0 0 0
21 23 1 0 0 0 0
21 22 1 0 0 0 0
24 25 1 0 0 0 0
24 54 1 1 0 0 0
25 55 2 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
29 56 2 0 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
16 38 1 1 0 0 0
17 39 1 1 0 0 0
43 42 1 0 0 0 0
42 45 1 0 0 0 0
42 44 2 0 0 0 0
39 42 1 0 0 0 0
2 3 1 0 0 0 0
3 4 1 0 0 0 0
4 37 2 0 0 0 0
4 33 1 0 0 0 0
1 35 1 0 0 0 0
1 34 2 0 0 0 0
2 1 1 0 0 0 0
2 36 1 6 0 0 0
S SKP 7
ID BMFYS4CAa020
NAME (2S) -4- [2- [3- [ [4- [ [ [(2R,3S,4R,5R) -5- (6-Aminopurin-9-yl) -4-hydroxy-3-phosphonooxyoxolan-2-yl] methoxy-hydroxyphosphoryl] oxy-hydroxyphosphoryl] oxy-2-hydroxy-3,3- dimethylbutanoyl] amino] propanoylamino] ethylsulfanyl] -2-hydroxy-4-oxobutanoic acid
CAS_RN 52691-41-5
FORMULA C25H40N7O20P3S
EXACTMASS 883.1261
AVERAGEMASS 883.6075
SMILES n(c3N)cnc(c32)n(cn2)[C@@H]([C@@H]1O)O[C@@H]([C@H]1OP(O)(O)=O)COP(OP(O)(=O)OCC([C@@H](O)C(NCCC(NCCSC(C[C@@H](C(O)=O)O)=O)=O)=O)(C)C)(O)=O
KEGG http://www.genome.jp/dbget-bin/www_bget?compound+C04348
M END