Mol:FL1CCAGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 34 0 0 0 0 0 0 0 0999 V2000 | + | 32 34 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5905 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5905 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5905 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5905 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0365 0.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0365 0.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4825 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4825 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4825 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4825 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0365 1.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0365 1.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0712 0.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0712 0.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6237 0.3706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6237 0.3706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1751 0.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1751 0.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7252 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7252 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2636 0.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2636 0.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8020 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8020 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8020 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8020 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2636 1.3024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2636 1.3024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7252 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7252 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0712 -0.5864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0712 -0.5864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0365 -0.5879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0365 -0.5879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0712 1.3307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0712 1.3307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3402 1.3023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3402 1.3023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9531 -0.8870 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.9531 -0.8870 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.6068 -1.3441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6068 -1.3441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1082 -1.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1082 -1.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.5886 -1.1559 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.5886 -1.1559 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.9767 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9767 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4860 -0.9782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.4860 -0.9782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.3982 -0.8480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3982 -0.8480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9337 -1.7743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9337 -1.7743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8226 -1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8226 -1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4632 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4632 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2109 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2109 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9478 1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9478 1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4479 2.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4479 2.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 10 1 0 0 0 0 | + | 15 10 1 0 0 0 0 |
| − | 7 16 2 0 0 0 0 | + | 7 16 2 0 0 0 0 |
| − | 3 17 1 0 0 0 0 | + | 3 17 1 0 0 0 0 |
| − | 5 18 1 0 0 0 0 | + | 5 18 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 17 1 0 0 0 0 | + | 23 17 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 29 30 | + | M SAL 2 2 29 30 |
| − | M SBL 2 1 31 | + | M SBL 2 1 31 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 31 -2.5331 -0.5797 | + | M SVB 2 31 -2.5331 -0.5797 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 31 32 | + | M SAL 1 2 31 32 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 -1.9478 1.6297 | + | M SVB 1 33 -1.9478 1.6297 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1CCAGS0001 | + | ID FL1CCAGS0001 |
| − | KNApSAcK_ID C00007885 | + | KNApSAcK_ID C00007885 |
| − | NAME Neosakuranin | + | NAME Neosakuranin |
| − | CAS_RN 31187-54-9 | + | CAS_RN 31187-54-9 |
| − | FORMULA C22H24O10 | + | FORMULA C22H24O10 |
| − | EXACTMASS 448.136946988 | + | EXACTMASS 448.136946988 |
| − | AVERAGEMASS 448.41996000000006 | + | AVERAGEMASS 448.41996000000006 |
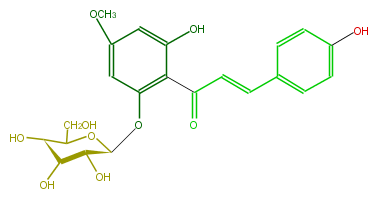
| − | SMILES O[C@@H]([C@H]3O)[C@@H](OC(CO)[C@@H]3O)Oc(c1)c(C(=O)C=Cc(c2)ccc(O)c2)c(cc1OC)O | + | SMILES O[C@@H]([C@H]3O)[C@@H](OC(CO)[C@@H]3O)Oc(c1)c(C(=O)C=Cc(c2)ccc(O)c2)c(cc1OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 34 0 0 0 0 0 0 0 0999 V2000
-1.5905 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5905 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0365 0.0514 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4825 0.3713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4825 1.0110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0365 1.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0712 0.0516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6237 0.3706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1751 0.0523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7252 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2636 0.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8020 0.3699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8020 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2636 1.3024 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7252 0.9916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0712 -0.5864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0365 -0.5879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0712 1.3307 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3402 1.3023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9531 -0.8870 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.6068 -1.3441 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1082 -1.1502 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.5886 -1.1559 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9767 -0.7953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4860 -0.9782 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.3982 -0.8480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9337 -1.7743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8226 -1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4632 -0.5776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2109 -0.2289 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9478 1.6297 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4479 2.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 10 1 0 0 0 0
7 16 2 0 0 0 0
3 17 1 0 0 0 0
5 18 1 0 0 0 0
13 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 17 1 0 0 0 0
25 29 1 0 0 0 0
29 30 1 0 0 0 0
1 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 29 30
M SBL 2 1 31
M SMT 2 CH2OH
M SVB 2 31 -2.5331 -0.5797
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 31 32
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 -1.9478 1.6297
S SKP 8
ID FL1CCAGS0001
KNApSAcK_ID C00007885
NAME Neosakuranin
CAS_RN 31187-54-9
FORMULA C22H24O10
EXACTMASS 448.136946988
AVERAGEMASS 448.41996000000006
SMILES O[C@@H]([C@H]3O)[C@@H](OC(CO)[C@@H]3O)Oc(c1)c(C(=O)C=Cc(c2)ccc(O)c2)c(cc1OC)O
M END