Mol:FL1DA9NC0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9165 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9165 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9165 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9165 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4508 0.5594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4508 0.5594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9850 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9850 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9850 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9850 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4508 1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4508 1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5194 0.5595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5194 0.5595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5194 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5194 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0679 -0.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0679 -0.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4309 0.2507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4309 0.2507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0679 0.7503 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.0679 0.7503 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.9419 -0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9419 -0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7873 -1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7873 -1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2101 -1.2124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2101 -1.2124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2124 -0.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2124 -0.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7888 -0.9445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7888 -0.9445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9430 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9430 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2109 -0.5227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2109 -0.5227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2109 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2109 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7369 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7369 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2381 0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2381 0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7394 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7394 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7394 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7394 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2381 1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2381 1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7369 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7369 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3821 0.5595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3821 0.5595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3821 0.0863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3821 0.0863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5194 1.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5194 1.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4001 1.0047 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4001 1.0047 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6150 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6150 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1916 1.2119 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1916 1.2119 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6150 1.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6150 1.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0999 1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0999 1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0873 0.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0873 0.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0915 -1.6551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0915 -1.6551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7394 1.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7394 1.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2395 2.0442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2395 2.0442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6527 -0.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6527 -0.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5176 -1.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5176 -1.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 7 1 0 0 0 0 | + | 11 7 1 0 0 0 0 |
| − | 8 12 2 0 0 0 0 | + | 8 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 9 2 0 0 0 0 | + | 15 9 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 16 18 1 0 0 0 0 | + | 16 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 2 26 1 0 0 0 0 | + | 2 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 5 28 1 0 0 0 0 | + | 5 28 1 0 0 0 0 |
| − | 7 29 1 1 0 0 0 | + | 7 29 1 1 0 0 0 |
| − | 11 30 1 1 0 0 0 | + | 11 30 1 1 0 0 0 |
| − | 11 31 1 6 0 0 0 | + | 11 31 1 6 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 14 35 1 0 0 0 0 | + | 14 35 1 0 0 0 0 |
| − | 26 36 1 0 0 0 0 | + | 26 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 12 38 1 0 0 0 0 | + | 12 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 41 -1.5243 0.0993 | + | M SVB 2 41 -1.5243 0.0993 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 36 37 | + | M SAL 1 2 36 37 |
| − | M SBL 1 1 39 | + | M SBL 1 1 39 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 39 -2.7394 1.1782 | + | M SVB 1 39 -2.7394 1.1782 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL1DA9NC0002 | + | ID FL1DA9NC0002 |
| − | KNApSAcK_ID C00000994 | + | KNApSAcK_ID C00000994 |
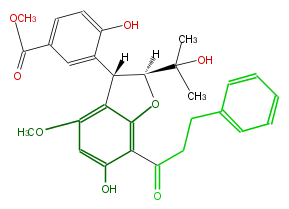
| − | NAME Piperaduncin B | + | NAME Piperaduncin B |
| − | CAS_RN 155023-55-5 | + | CAS_RN 155023-55-5 |
| − | FORMULA C29H30O8 | + | FORMULA C29H30O8 |
| − | EXACTMASS 506.194067936 | + | EXACTMASS 506.194067936 |
| − | AVERAGEMASS 506.5437 | + | AVERAGEMASS 506.5437 |
| − | SMILES [C@@](C(C)(C)O)(O2)([C@]([H])(c(c4OC)c2c(c(c4)O)C(=O)CCc(c3)cccc3)c(c1)c(ccc1C(OC)=O)O)[H] | + | SMILES [C@@](C(C)(C)O)(O2)([C@]([H])(c(c4OC)c2c(c(c4)O)C(=O)CCc(c3)cccc3)c(c1)c(ccc1C(OC)=O)O)[H] |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
-1.9165 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9165 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4508 0.5594 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9850 0.8283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9850 1.3661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4508 1.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5194 0.5595 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5194 -0.0580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0679 -0.2489 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4309 0.2507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0679 0.7503 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.9419 -0.4805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7873 -1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2101 -1.2124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2124 -0.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7888 -0.9445 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9430 -1.5210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2109 -0.5227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2109 0.1295 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7369 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2381 0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7394 0.4332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7394 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2381 1.3014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7369 1.0120 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3821 0.5595 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3821 0.0863 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5194 1.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4001 1.0047 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
0.6150 1.0662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1916 1.2119 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
0.6150 1.6551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0999 1.3461 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0873 0.5939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0915 -1.6551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7394 1.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2395 2.0442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6527 -0.8930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5176 -1.3949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 1 0 0 0 0
11 7 1 0 0 0 0
8 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 9 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
16 18 1 0 0 0 0
18 19 1 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 20 1 0 0 0 0
2 26 1 0 0 0 0
26 27 2 0 0 0 0
5 28 1 0 0 0 0
7 29 1 1 0 0 0
11 30 1 1 0 0 0
11 31 1 6 0 0 0
30 32 1 0 0 0 0
30 33 1 0 0 0 0
30 34 1 0 0 0 0
14 35 1 0 0 0 0
26 36 1 0 0 0 0
36 37 1 0 0 0 0
12 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 41
M SMT 2 OCH3
M SVB 2 41 -1.5243 0.0993
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 36 37
M SBL 1 1 39
M SMT 1 OCH3
M SVB 1 39 -2.7394 1.1782
S SKP 8
ID FL1DA9NC0002
KNApSAcK_ID C00000994
NAME Piperaduncin B
CAS_RN 155023-55-5
FORMULA C29H30O8
EXACTMASS 506.194067936
AVERAGEMASS 506.5437
SMILES [C@@](C(C)(C)O)(O2)([C@]([H])(c(c4OC)c2c(c(c4)O)C(=O)CCc(c3)cccc3)c(c1)c(ccc1C(OC)=O)O)[H]
M END