Mol:FL2F1CGS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.4438 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4438 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4438 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4438 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0566 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0566 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5571 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5571 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5571 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5571 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0566 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0566 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0575 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0575 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5580 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5580 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5580 -0.6766 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.5580 -0.6766 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.0575 -0.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0575 -0.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0581 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0581 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5724 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5724 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0868 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0868 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0868 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0868 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5724 0.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5724 0.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0581 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0581 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5580 0.0040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5580 0.0040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9440 -0.3878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9440 -0.3878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4555 -0.6832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.4555 -0.6832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -3.2282 -0.5941 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.2282 -0.5941 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.7631 -1.0592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.7631 -1.0592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.1916 -0.7335 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.1916 -0.7335 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.9990 -0.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9990 -0.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5706 -0.5941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.5706 -0.5941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.7380 -1.1871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7380 -1.1871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7459 -0.6854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7459 -0.6854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0457 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0457 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6000 0.5024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6000 0.5024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2942 1.3859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2942 1.3859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7826 1.0906 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.7826 1.0906 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.0099 1.1797 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.0099 1.1797 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.4750 0.7146 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4750 0.7146 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0465 1.0403 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.0465 1.0403 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.2391 1.5054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2391 1.5054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6676 1.1797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.6676 1.1797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.5001 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5001 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3929 1.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3929 1.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1708 0.5415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1708 0.5415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0575 -1.9823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0575 -1.9823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2797 -0.0162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2797 -0.0162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5618 0.7591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5618 0.7591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2691 1.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2691 1.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8224 2.8153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8224 2.8153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 9 17 1 1 0 0 0 | + | 9 17 1 1 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 19 22 1 1 0 0 0 | + | 19 22 1 1 0 0 0 |
| − | 19 23 1 0 0 0 0 | + | 19 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 20 1 0 0 0 0 | + | 24 20 1 0 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 14 28 1 0 0 0 0 | + | 14 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 30 33 1 1 0 0 0 | + | 30 33 1 1 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 31 1 0 0 0 0 | + | 35 31 1 0 0 0 0 |
| − | 33 36 1 0 0 0 0 | + | 33 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 7 39 2 0 0 0 0 | + | 7 39 2 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 0.2691 1.9823 | + | M SVB 2 46 0.2691 1.9823 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.969 0.2293 | + | M SVB 1 44 -2.969 0.2293 |
| − | S SKP 8 | + | S SKP 8 |
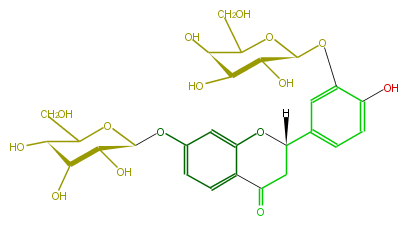
| − | ID FL2F1CGS0001 | + | ID FL2F1CGS0001 |
| − | KNApSAcK_ID C00000946 | + | KNApSAcK_ID C00000946 |
| − | NAME Butrin | + | NAME Butrin |
| − | CAS_RN 492-13-7 | + | CAS_RN 492-13-7 |
| − | FORMULA C27H32O15 | + | FORMULA C27H32O15 |
| − | EXACTMASS 596.174120354 | + | EXACTMASS 596.174120354 |
| − | AVERAGEMASS 596.5339799999999 | + | AVERAGEMASS 596.5339799999999 |
| − | SMILES c(c1)c(O[C@@H]([C@@H](O)5)OC([C@H](O)[C@H](O)5)CO)cc(O2)c1C(C[C@@](c(c3)ccc(O)c(O[C@H](O4)[C@@H](O)[C@H]([C@@H](O)C(CO)4)O)3)2[H])=O | + | SMILES c(c1)c(O[C@@H]([C@@H](O)5)OC([C@H](O)[C@H](O)5)CO)cc(O2)c1C(C[C@@](c(c3)ccc(O)c(O[C@H](O4)[C@@H](O)[C@H]([C@@H](O)C(CO)4)O)3)2[H])=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.4438 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4438 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0566 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5571 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5571 -0.6766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0566 -0.3877 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0575 -1.5434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5580 -1.2545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5580 -0.6766 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.0575 -0.3877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0581 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5724 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0868 -0.3878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0868 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5724 0.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0581 0.2061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5580 0.0040 0.0000 H 0 0 0 0 0 0 0 0 0 0 0 0
-0.9440 -0.3878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4555 -0.6832 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-3.2282 -0.5941 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.7631 -1.0592 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.1916 -0.7335 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.9990 -0.2683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5706 -0.5941 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.7380 -1.1871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7459 -0.6854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0457 -1.6650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6000 0.5024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2942 1.3859 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7826 1.0906 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.0099 1.1797 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.4750 0.7146 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0465 1.0403 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.2391 1.5054 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6676 1.1797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.5001 0.5866 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3929 1.5176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1708 0.5415 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0575 -1.9823 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2797 -0.0162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5618 0.7591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2691 1.9823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8224 2.8153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
9 17 1 1 0 0 0
1 18 1 0 0 0 0
18 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
19 22 1 1 0 0 0
19 23 1 0 0 0 0
23 24 1 0 0 0 0
24 20 1 0 0 0 0
22 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
14 28 1 0 0 0 0
29 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
30 33 1 1 0 0 0
30 34 1 0 0 0 0
34 35 1 0 0 0 0
35 31 1 0 0 0 0
33 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
15 29 1 0 0 0 0
7 39 2 0 0 0 0
24 40 1 0 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 0.2691 1.9823
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.969 0.2293
S SKP 8
ID FL2F1CGS0001
KNApSAcK_ID C00000946
NAME Butrin
CAS_RN 492-13-7
FORMULA C27H32O15
EXACTMASS 596.174120354
AVERAGEMASS 596.5339799999999
SMILES c(c1)c(O[C@@H]([C@@H](O)5)OC([C@H](O)[C@H](O)5)CO)cc(O2)c1C(C[C@@](c(c3)ccc(O)c(O[C@H](O4)[C@@H](O)[C@H]([C@@H](O)C(CO)4)O)3)2[H])=O
M END