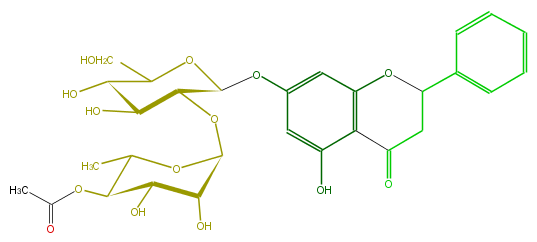
Mol:FL2FA9GS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2681 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2681 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2682 -0.2157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2682 -0.2157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9826 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9826 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6970 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6970 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6969 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6969 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9826 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9826 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4113 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4113 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1258 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1258 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1258 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1258 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4113 1.0217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4113 1.0217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8398 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8398 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5680 0.6011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5680 0.6011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2961 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2961 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2961 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2961 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5680 2.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5680 2.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8399 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8399 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4113 -1.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4113 -1.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3842 0.9801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3842 0.9801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9826 -1.4529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9826 -1.4529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1290 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1290 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7927 1.2935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7927 1.2935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6082 0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6082 0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5466 0.8287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5466 0.8287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8830 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8830 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0675 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0675 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7632 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7632 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2514 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2514 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2780 0.0506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2780 0.0506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6180 -1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6180 -1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1201 -0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1201 -0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0778 -1.0144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0778 -1.0144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0415 -0.7405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0415 -0.7405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5397 -1.6031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5397 -1.6031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5818 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5818 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7763 -0.9375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7763 -0.9375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5696 -2.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5696 -2.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9652 -1.9249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9652 -1.9249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1478 -1.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1478 -1.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7390 -1.7817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7390 -1.7817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2961 -1.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2961 -1.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7391 -2.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7391 -2.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3676 1.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3676 1.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4286 2.1765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4286 2.1765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 20 25 1 1 0 0 0 | + | 20 25 1 1 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 29 34 1 1 0 0 0 | + | 29 34 1 1 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 32 35 1 0 0 0 0 | + | 32 35 1 0 0 0 0 |
| − | 29 36 1 0 0 0 0 | + | 29 36 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 39 41 2 0 0 0 0 | + | 39 41 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 22 42 1 0 0 0 0 | + | 22 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 47 0.7594 -0.4716 | + | M SBV 1 47 0.7594 -0.4716 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FA9GS0006 | + | ID FL2FA9GS0006 |
| − | FORMULA C29H34O14 | + | FORMULA C29H34O14 |
| − | EXACTMASS 606.194855796 | + | EXACTMASS 606.194855796 |
| − | AVERAGEMASS 606.57186 | + | AVERAGEMASS 606.57186 |
| − | SMILES C(C(CO)2)(O)C(O)C(C(Oc(c5)cc(O3)c(c(O)5)C(CC(c(c4)cccc4)3)=O)O2)OC(C(O)1)OC(C(OC(C)=O)C1O)C | + | SMILES C(C(CO)2)(O)C(O)C(C(Oc(c5)cc(O3)c(c(O)5)C(CC(c(c4)cccc4)3)=O)O2)OC(C(O)1)OC(C(OC(C)=O)C1O)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.2681 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2682 -0.2157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9826 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6970 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6969 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9826 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4113 -0.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1258 -0.2158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1258 0.6092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4113 1.0217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8398 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5680 0.6011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2961 1.0214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2961 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5680 2.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8399 1.8622 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4113 -1.3265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3842 0.9801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9826 -1.4529 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1290 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7927 1.2935 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6082 0.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5466 0.8287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8830 0.1650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0675 0.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7632 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2514 0.5872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2780 0.0506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6180 -1.6033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1201 -0.7407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0778 -1.0144 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0415 -0.7405 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5397 -1.6031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5818 -1.3296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7763 -0.9375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5696 -2.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9652 -1.9249 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1478 -1.4404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7390 -1.7817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2961 -1.4599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7391 -2.2827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3676 1.3004 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4286 2.1765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
20 25 1 1 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
25 28 1 0 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
29 34 1 1 0 0 0
28 30 1 0 0 0 0
32 35 1 0 0 0 0
29 36 1 0 0 0 0
34 37 1 0 0 0 0
33 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
39 41 2 0 0 0 0
42 43 1 0 0 0 0
22 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 ^ CH2OH
M SBV 1 47 0.7594 -0.4716
S SKP 5
ID FL2FA9GS0006
FORMULA C29H34O14
EXACTMASS 606.194855796
AVERAGEMASS 606.57186
SMILES C(C(CO)2)(O)C(O)C(C(Oc(c5)cc(O3)c(c(O)5)C(CC(c(c4)cccc4)3)=O)O2)OC(C(O)1)OC(C(OC(C)=O)C1O)C
M END