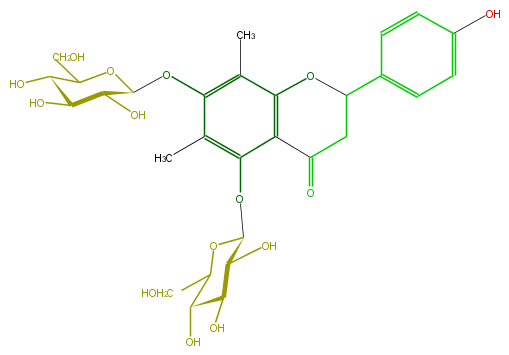
Mol:FL2FAAGM0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7108 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7108 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7108 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7108 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0037 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0037 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7182 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7182 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7182 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7182 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0037 2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0037 2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4327 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4327 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1472 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1472 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1472 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1472 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4327 2.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4327 2.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8615 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8615 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5898 1.7834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5898 1.7834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3180 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3180 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3180 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3180 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5898 3.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5898 3.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8615 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8615 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4327 -0.1445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4327 -0.1445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4707 2.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4707 2.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0037 -0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0037 -0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4251 0.5541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4251 0.5541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0460 3.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0460 3.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0037 3.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0037 3.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8408 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8408 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3988 1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3988 1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7624 1.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7624 1.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1483 1.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1483 1.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5945 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5945 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2447 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2447 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4284 2.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4284 2.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0210 1.6689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0210 1.6689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1283 1.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1283 1.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9911 -2.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9911 -2.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3281 -2.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3281 -2.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2434 -1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2434 -1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0772 -1.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0772 -1.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5326 -1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5326 -1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5922 -1.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5922 -1.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0026 -3.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0026 -3.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5183 -2.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5183 -2.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5309 -1.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5309 -1.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8031 2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8031 2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0460 2.9153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0460 2.9153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2771 -2.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2771 -2.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2771 -3.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2771 -3.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 2 20 1 0 0 0 0 | + | 2 20 1 0 0 0 0 |
| − | 14 21 1 0 0 0 0 | + | 14 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 18 26 1 0 0 0 0 | + | 18 26 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 19 1 0 0 0 0 | + | 35 19 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 28 41 1 0 0 0 0 | + | 28 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.5584 -0.5237 | + | M SBV 1 46 0.5584 -0.5237 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 48 0.6849 0.3741 | + | M SBV 2 48 0.6849 0.3741 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL2FAAGM0003 | + | ID FL2FAAGM0003 |
| − | FORMULA C29H36O15 | + | FORMULA C29H36O15 |
| − | EXACTMASS 624.2054204819999 | + | EXACTMASS 624.2054204819999 |
| − | AVERAGEMASS 624.58714 | + | AVERAGEMASS 624.58714 |
| − | SMILES OC(C(O)5)C(CO)OC(C5O)Oc(c13)c(c(OC(O4)C(C(C(O)C4CO)O)O)c(c1OC(CC3=O)c(c2)ccc(c2)O)C)C | + | SMILES OC(C(O)5)C(CO)OC(C5O)Oc(c13)c(c(OC(O4)C(C(C(O)C4CO)O)O)c(c1OC(CC3=O)c(c2)ccc(c2)O)C)C |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.7108 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7108 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0037 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7182 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7182 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0037 2.2041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4327 0.5540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1472 0.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1472 1.7915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4327 2.2041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8615 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5898 1.7834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3180 2.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3180 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5898 3.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8615 3.0448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4327 -0.1445 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4707 2.2303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0037 -0.2709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4251 0.5541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0460 3.4651 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0037 3.0288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8408 2.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3988 1.6007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7624 1.8482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1483 1.8549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5945 2.3012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2447 2.0677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4284 2.0469 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0210 1.6689 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1283 1.4203 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9911 -2.4711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3281 -2.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2434 -1.6019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0772 -1.0655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5326 -1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5922 -1.8067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0026 -3.1602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5183 -2.8837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5309 -1.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8031 2.5915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0460 2.9153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2771 -2.1808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2771 -3.4652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
2 20 1 0 0 0 0
14 21 1 0 0 0 0
6 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
18 26 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 19 1 0 0 0 0
41 42 1 0 0 0 0
28 41 1 0 0 0 0
43 44 1 0 0 0 0
37 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.5584 -0.5237
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 ^ CH2OH
M SBV 2 48 0.6849 0.3741
S SKP 5
ID FL2FAAGM0003
FORMULA C29H36O15
EXACTMASS 624.2054204819999
AVERAGEMASS 624.58714
SMILES OC(C(O)5)C(CO)OC(C5O)Oc(c13)c(c(OC(O4)C(C(C(O)C4CO)O)O)c(c1OC(CC3=O)c(c2)ccc(c2)O)C)C
M END