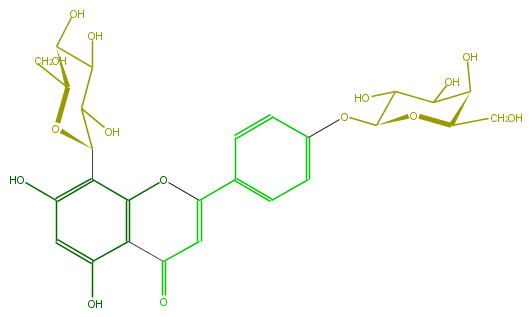
Mol:FL3FAADS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.0162 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0162 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0162 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0162 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3017 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3017 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5873 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5873 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5873 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5873 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3017 -0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3017 -0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8728 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8728 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1584 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1584 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1584 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1584 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8728 -0.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8728 -0.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8728 -2.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8728 -2.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4442 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4442 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2840 -0.9378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2840 -0.9378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0121 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0121 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0121 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0121 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2840 0.7439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2840 0.7439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4442 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4442 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7402 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7402 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3017 -2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3017 -2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7304 -0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7304 -0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0264 0.5094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0264 0.5094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2971 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2971 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5285 0.8982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5285 0.8982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2971 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2971 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0264 2.1341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0264 2.1341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7951 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7951 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6772 2.8194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6772 2.8194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3036 2.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3036 2.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9702 0.4428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9702 0.4428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7691 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7691 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3827 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3827 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1257 0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1257 0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8732 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8732 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2596 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2596 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5165 1.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5165 1.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1766 1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1766 1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8394 1.4441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8394 1.4441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1991 1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1991 1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7304 0.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7304 0.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7304 -0.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7304 -0.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4894 1.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4894 1.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8473 2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8473 2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 26 21 1 1 0 0 0 | + | 26 21 1 1 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 22 6 1 0 0 0 0 | + | 22 6 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 31 18 1 0 0 0 0 | + | 31 18 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -0.8572 -0.1581 | + | M SBV 1 44 -0.8572 -0.1581 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 46 0.6943 -0.5555 | + | M SBV 2 46 0.6943 -0.5555 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FAADS0002 | + | ID FL3FAADS0002 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O | + | SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-4.0162 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0162 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3017 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5873 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5873 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3017 -0.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8728 -2.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1584 -1.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1584 -0.9297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8728 -0.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8728 -2.9525 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4442 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2840 -0.9378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0121 -0.5174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0121 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2840 0.7439 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4442 0.3234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7402 0.7438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3017 -2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7304 -0.5174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0264 0.5094 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2971 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5285 0.8982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2971 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0264 2.1341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7951 1.3242 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6772 2.8194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3036 2.3688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9702 0.4428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7691 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3827 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1257 0.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8732 0.5648 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2596 1.2339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5165 1.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1766 1.1228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8394 1.4441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1991 1.9460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7304 0.7229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7304 -0.3367 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4894 1.8797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8473 2.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
3 19 1 0 0 0 0
1 20 1 0 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
26 21 1 1 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
23 29 1 0 0 0 0
22 6 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
31 18 1 0 0 0 0
39 40 1 0 0 0 0
33 39 1 0 0 0 0
41 42 1 0 0 0 0
26 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -0.8572 -0.1581
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 ^ CH2OH
M SBV 2 46 0.6943 -0.5555
S SKP 5
ID FL3FAADS0002
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5)(ccc(c5)OC(O4)C(O)C(O)C(O)C4CO)C(O3)=CC(=O)c(c13)c(O)cc(c(C(C(O)2)OC(C(O)C(O)2)CO)1)O
M END