Mol:FL3FAADS0046
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.1253 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1253 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4108 0.9517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4108 0.9517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6963 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6963 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6963 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6963 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4108 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4108 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1253 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1253 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2674 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2674 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4108 -1.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4108 -1.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9266 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9266 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6411 0.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6411 0.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3556 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3556 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3556 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3556 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6411 2.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6411 2.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9266 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9266 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0390 2.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0390 2.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9819 -1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9819 -1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5769 0.9378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5769 0.9378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4721 0.0281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4721 0.0281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0594 -0.6866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0594 -0.6866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2611 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2611 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5324 -0.7043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5324 -0.7043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1198 0.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1198 0.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6786 -0.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6786 -0.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8356 0.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8356 0.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9553 -0.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9553 -0.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5081 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5081 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4326 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4326 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2964 0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2964 0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0726 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0726 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6599 -1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6599 -1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8616 -1.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8616 -1.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0681 -1.8263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0681 -1.8263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4807 -1.1115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4807 -1.1115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2791 -1.3206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2791 -1.3206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4361 -0.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4361 -0.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9159 -0.4577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9159 -0.4577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5558 -1.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5558 -1.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1086 -1.5495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1086 -1.5495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0331 -2.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0331 -2.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3771 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3771 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9651 0.9846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9651 0.9846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2010 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2010 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6129 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6129 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4368 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4368 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8493 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8493 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.6743 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.6743 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.0868 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.0868 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.6743 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.6743 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8493 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8493 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.9107 -0.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.9107 -0.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.4564 -0.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.4564 -0.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2742 -0.3281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2742 -0.3281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.3836 0.4899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.3836 0.4899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.8946 1.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.8946 1.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.0767 1.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.0767 1.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.9673 0.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.9673 0.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3657 0.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3657 0.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9273 -0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9273 -0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7202 -1.0691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7202 -1.0691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8643 -0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8643 -0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.9107 0.0885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.9107 0.0885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 5 11 2 0 0 0 0 | + | 5 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 15 18 1 0 0 0 0 | + | 15 18 1 0 0 0 0 |
| − | 10 19 1 0 0 0 0 | + | 10 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 9 1 0 0 0 0 | + | 24 9 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 54 62 1 0 0 0 0 | + | 54 62 1 0 0 0 0 |
| − | 55 63 1 0 0 0 0 | + | 55 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 18 1 0 0 0 0 | + | 57 18 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAADS0046 | + | ID FL3FAADS0046 |
| − | KNApSAcK_ID C00014081 | + | KNApSAcK_ID C00014081 |
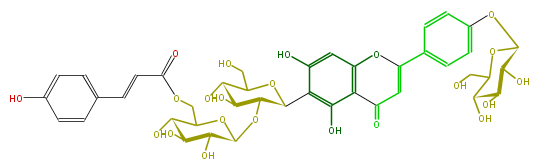
| − | NAME Isovitexin 2''-O-(6'''-(E)-p-coumaroyl)glucoside 4'-O-glucoside | + | NAME Isovitexin 2''-O-(6'''-(E)-p-coumaroyl)glucoside 4'-O-glucoside |
| − | CAS_RN 372113-51-4 | + | CAS_RN 372113-51-4 |
| − | FORMULA C42H46O22 | + | FORMULA C42H46O22 |
| − | EXACTMASS 902.248073156 | + | EXACTMASS 902.248073156 |
| − | AVERAGEMASS 902.80144 | + | AVERAGEMASS 902.80144 |
| − | SMILES c(C7=O)(c(O)1)c(OC(=C7)c(c6)ccc(c6)OC(O5)C(O)C(O)C(C5CO)O)cc(c1C(C(OC(C(O)3)OC(COC(=O)C=Cc(c4)ccc(O)c4)C(O)C3O)2)OC(C(C2O)O)CO)O | + | SMILES c(C7=O)(c(O)1)c(OC(=C7)c(c6)ccc(c6)OC(O5)C(O)C(O)C(C5CO)O)cc(c1C(C(OC(C(O)3)OC(COC(=O)C=Cc(c4)ccc(O)c4)C(O)C3O)2)OC(C(C2O)O)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
4.1253 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4108 0.9517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6963 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6963 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4108 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1253 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 0.9517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 0.5392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2674 -0.2858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 -0.6983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4108 -1.2704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9266 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6411 0.5894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3556 1.0019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3556 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6411 2.2394 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9266 1.8269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0390 2.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9819 -1.4683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5769 0.9378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4721 0.0281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0594 -0.6866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2611 -0.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5324 -0.7043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1198 0.0105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6786 -0.1986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8356 0.3873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9553 -0.4551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5081 -0.4275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4326 -1.1174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2964 0.6533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0726 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6599 -1.8086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8616 -1.5994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0681 -1.8263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4807 -1.1115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2791 -1.3206 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4361 -0.7347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9159 -0.4577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5558 -1.5771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1086 -1.5495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0331 -2.2394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3771 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9651 0.9846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2010 0.2711 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6129 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4368 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8493 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.6743 0.2720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.0868 -0.4424 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.6743 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8493 -1.1569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.9107 -0.4424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.4564 -0.4387 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.2742 -0.3281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.3836 0.4899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.8946 1.1380 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
7.0767 1.0275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.9673 0.2095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.3657 0.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9273 -0.0524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.7202 -1.0691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8643 -0.6449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.9107 0.0885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
5 11 2 0 0 0 0
1 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
15 18 1 0 0 0 0
10 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 31 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 9 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 30 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
39 43 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 54 1 0 0 0 0
59 60 1 0 0 0 0
60 61 1 0 0 0 0
54 62 1 0 0 0 0
55 63 1 0 0 0 0
56 64 1 0 0 0 0
57 18 1 0 0 0 0
S SKP 8
ID FL3FAADS0046
KNApSAcK_ID C00014081
NAME Isovitexin 2''-O-(6'''-(E)-p-coumaroyl)glucoside 4'-O-glucoside
CAS_RN 372113-51-4
FORMULA C42H46O22
EXACTMASS 902.248073156
AVERAGEMASS 902.80144
SMILES c(C7=O)(c(O)1)c(OC(=C7)c(c6)ccc(c6)OC(O5)C(O)C(O)C(C5CO)O)cc(c1C(C(OC(C(O)3)OC(COC(=O)C=Cc(c4)ccc(O)c4)C(O)C3O)2)OC(C(C2O)O)CO)O
M END