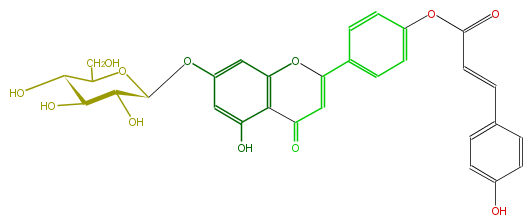
Mol:FL3FAAGN0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1562 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1562 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1562 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1562 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5999 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5999 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0436 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0436 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0436 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0436 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5999 1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5999 1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5127 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5127 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0690 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0690 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0690 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0690 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5127 1.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5127 1.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5127 -0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5127 -0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6251 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6251 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1920 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1920 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7590 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7590 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7590 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7590 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1920 2.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1920 2.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6251 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6251 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7123 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7123 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3258 2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3258 2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5999 -0.7282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5999 -0.7282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2724 0.7398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2724 0.7398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8377 0.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8377 0.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2118 0.4094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2118 0.4094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5253 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5253 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0467 0.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0467 0.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6861 0.6253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6861 0.6253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1608 0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1608 0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5153 0.1330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5153 0.1330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8532 -0.1926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8532 -0.1926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9829 1.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9829 1.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6389 2.0390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6389 2.0390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9829 0.9025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9829 0.9025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6390 0.5237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6390 0.5237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6391 -0.2325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6391 -0.2325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1174 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1174 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1174 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1174 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6391 -1.4373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6391 -1.4373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1608 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1608 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1608 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1608 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6391 -2.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6391 -2.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7226 1.0238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7226 1.0238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5195 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5195 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 18 24 1 0 0 0 0 | + | 18 24 1 0 0 0 0 |
| − | 19 30 1 0 0 0 0 | + | 19 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 26 41 1 0 0 0 0 | + | 26 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 45 -6.2417 4.1049 | + | M SBV 1 45 -6.2417 4.1049 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAAGN0001 | + | ID FL3FAAGN0001 |
| − | KNApSAcK_ID C00004183 | + | KNApSAcK_ID C00004183 |
| − | NAME Apigenin 7-glucoside-4'-p-coumarate | + | NAME Apigenin 7-glucoside-4'-p-coumarate |
| − | CAS_RN 61237-20-5 | + | CAS_RN 61237-20-5 |
| − | FORMULA C30H26O12 | + | FORMULA C30H26O12 |
| − | EXACTMASS 578.1424262959999 | + | EXACTMASS 578.1424262959999 |
| − | AVERAGEMASS 578.5202400000001 | + | AVERAGEMASS 578.5202400000001 |
| − | SMILES c(C=CC(Oc(c2)ccc(C(O5)=CC(c(c53)c(O)cc(OC(C4O)OC(C(O)C4O)CO)c3)=O)c2)=O)(c1)ccc(c1)O | + | SMILES c(C=CC(Oc(c2)ccc(C(O5)=CC(c(c53)c(O)cc(OC(C4O)OC(C(O)C4O)CO)c3)=O)c2)=O)(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-1.1562 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1562 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5999 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0436 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0436 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5999 1.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5127 -0.2271 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0690 0.0941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0690 0.7365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5127 1.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5127 -0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6251 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1920 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7590 1.0575 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7590 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1920 2.0396 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6251 1.7122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7123 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3258 2.0395 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5999 -0.7282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2724 0.7398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8377 0.1660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2118 0.4094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5253 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0467 0.8549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6861 0.6253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1608 0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5153 0.1330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8532 -0.1926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9829 1.6601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6389 2.0390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9829 0.9025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6390 0.5237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6391 -0.2325 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1174 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1174 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6391 -1.4373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1608 -1.1361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1608 -0.5337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6391 -2.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7226 1.0238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5195 1.2373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
18 24 1 0 0 0 0
19 30 1 0 0 0 0
30 31 2 0 0 0 0
30 32 1 0 0 0 0
32 33 2 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
26 41 1 0 0 0 0
41 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 45
M SMT 1 ^CH2OH
M SBV 1 45 -6.2417 4.1049
S SKP 8
ID FL3FAAGN0001
KNApSAcK_ID C00004183
NAME Apigenin 7-glucoside-4'-p-coumarate
CAS_RN 61237-20-5
FORMULA C30H26O12
EXACTMASS 578.1424262959999
AVERAGEMASS 578.5202400000001
SMILES c(C=CC(Oc(c2)ccc(C(O5)=CC(c(c53)c(O)cc(OC(C4O)OC(C(O)C4O)CO)c3)=O)c2)=O)(c1)ccc(c1)O
M END