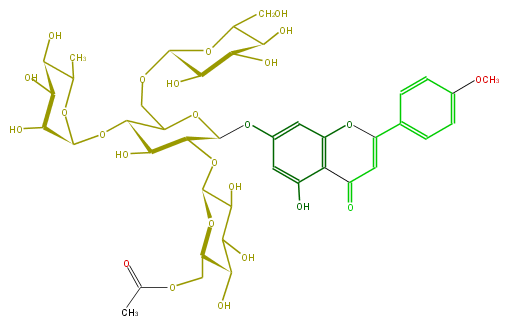
Mol:FL3FABGS0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1196 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1196 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1195 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1195 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8077 -1.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8077 -1.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4959 -0.7753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4959 -0.7753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4958 0.0193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4958 0.0193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8077 0.4167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8077 0.4167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1838 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1838 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8720 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8720 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8720 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8720 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1839 0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1839 0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1937 -1.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1937 -1.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5599 0.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5599 0.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2611 0.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2611 0.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9624 0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9624 0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9624 1.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9624 1.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2611 1.6313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2611 1.6313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5599 1.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5599 1.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5683 0.4165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5683 0.4165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8171 -1.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8171 -1.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8367 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8367 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1813 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1813 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2374 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2374 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3266 0.0101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3266 0.0101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9883 0.6720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9883 0.6720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8142 0.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8142 0.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4388 0.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4388 0.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4890 0.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4890 0.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8789 -0.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8789 -0.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4644 -0.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4644 -0.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6873 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6873 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8302 1.7060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8302 1.7060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0136 2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0136 2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1363 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1363 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9471 3.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9471 3.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6319 2.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6319 2.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4283 1.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4283 1.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5032 1.5072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5032 1.5072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0468 2.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0468 2.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3554 2.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3554 2.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8088 -3.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8088 -3.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0102 -3.6221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0102 -3.6221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2635 -2.7354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2635 -2.7354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0102 -1.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0102 -1.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8088 -1.3822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8088 -1.3822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5555 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5555 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0102 -1.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0102 -1.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8072 -3.7630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8072 -3.7630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6108 -4.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6108 -4.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4813 -4.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4813 -4.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8625 -3.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8625 -3.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9106 -4.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9106 -4.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2984 -4.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2984 -4.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6542 -3.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6542 -3.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0804 0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0804 0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2818 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2818 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5352 0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5352 0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2818 1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2818 1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0804 2.0811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0804 2.0811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8270 1.1944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8270 1.1944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3032 2.2169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3032 2.2169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.5208 1.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.5208 1.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7535 0.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7535 0.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8861 2.8064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8861 2.8064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2144 3.4143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2144 3.4143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0243 4.6630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0243 4.6630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6635 1.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6635 1.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.7535 1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.7535 1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 20 27 1 0 0 0 0 | + | 20 27 1 0 0 0 0 |
| − | 21 28 1 0 0 0 0 | + | 21 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 36 26 1 0 0 0 0 | + | 36 26 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 40 1 1 0 0 0 | + | 45 40 1 1 0 0 0 |
| − | 43 46 1 0 0 0 0 | + | 43 46 1 0 0 0 0 |
| − | 44 29 1 0 0 0 0 | + | 44 29 1 0 0 0 0 |
| − | 40 47 1 0 0 0 0 | + | 40 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 41 52 1 0 0 0 0 | + | 41 52 1 0 0 0 0 |
| − | 42 53 1 0 0 0 0 | + | 42 53 1 0 0 0 0 |
| − | 55 54 1 1 0 0 0 | + | 55 54 1 1 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 1 0 0 0 | + | 58 59 1 1 0 0 0 |
| − | 59 54 1 1 0 0 0 | + | 59 54 1 1 0 0 0 |
| − | 57 60 1 0 0 0 0 | + | 57 60 1 0 0 0 0 |
| − | 59 61 1 0 0 0 0 | + | 59 61 1 0 0 0 0 |
| − | 54 62 1 0 0 0 0 | + | 54 62 1 0 0 0 0 |
| − | 55 27 1 0 0 0 0 | + | 55 27 1 0 0 0 0 |
| − | 58 63 1 0 0 0 0 | + | 58 63 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 34 64 1 0 0 0 0 | + | 34 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 15 66 1 0 0 0 0 | + | 15 66 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 64 65 | + | M SAL 1 2 64 65 |
| − | M SBL 1 1 71 | + | M SBL 1 1 71 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 71 -0.7327 -0.3960 | + | M SBV 1 71 -0.7327 -0.3960 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 66 67 | + | M SAL 2 2 66 67 |
| − | M SBL 2 1 73 | + | M SBL 2 1 73 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 73 -0.7011 -0.4047 | + | M SBV 2 73 -0.7011 -0.4047 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FABGS0012 | + | ID FL3FABGS0012 |
| − | FORMULA C42H54O25 | + | FORMULA C42H54O25 |
| − | EXACTMASS 958.2954172780001 | + | EXACTMASS 958.2954172780001 |
| − | AVERAGEMASS 958.86316 | + | AVERAGEMASS 958.86316 |
| − | SMILES c(O6)(c(C(C=C6c(c7)ccc(c7)OC)=O)5)cc(cc(O)5)OC(O1)C(OC(C4O)OC(COC(C)=O)C(O)C4O)C(C(OC(C(O)3)OC(C(C(O)3)O)C)C1COC(C2O)OC(C(O)C2O)CO)O | + | SMILES c(O6)(c(C(C=C6c(c7)ccc(c7)OC)=O)5)cc(cc(O)5)OC(O1)C(OC(C4O)OC(COC(C)=O)C(O)C4O)C(C(OC(C(O)3)OC(C(C(O)3)O)C)C1COC(C2O)OC(C(O)C2O)CO)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
0.1196 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1195 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8077 -1.1725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4959 -0.7753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4958 0.0193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8077 0.4167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1838 -1.1724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8720 -0.7752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8720 0.0194 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1839 0.4166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1937 -1.8506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5599 0.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2611 0.0116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9624 0.4166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9624 1.2264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2611 1.6313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5599 1.2263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5683 0.4165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8171 -1.8073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8367 0.4984 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1813 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2374 0.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3266 0.0101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9883 0.6720 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8142 0.2362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4388 0.8924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4890 0.1344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8789 -0.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4644 -0.6325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6873 2.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8302 1.7060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0136 2.3049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1363 2.5502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9471 3.0183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6319 2.3835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4283 1.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5032 1.5072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0468 2.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3554 2.9468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8088 -3.1610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0102 -3.6221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2635 -2.7354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0102 -1.8434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8088 -1.3822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5555 -2.2688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0102 -1.2679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8072 -3.7630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6108 -4.0091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4813 -4.0091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8625 -3.3487 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9106 -4.6630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2984 -4.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6542 -3.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0804 0.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2818 -0.1588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5352 0.7279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2818 1.6199 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0804 2.0811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8270 1.1944 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3032 2.2169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.5208 1.6850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.7535 0.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.8861 2.8064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2144 3.4143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0243 4.6630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6635 1.6312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.7535 1.0020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
25 26 1 0 0 0 0
20 27 1 0 0 0 0
21 28 1 0 0 0 0
22 29 1 0 0 0 0
23 18 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
36 26 1 0 0 0 0
33 39 1 0 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 1 0 0 0
45 40 1 1 0 0 0
43 46 1 0 0 0 0
44 29 1 0 0 0 0
40 47 1 0 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
41 52 1 0 0 0 0
42 53 1 0 0 0 0
55 54 1 1 0 0 0
55 56 1 0 0 0 0
56 57 1 0 0 0 0
57 58 1 0 0 0 0
58 59 1 1 0 0 0
59 54 1 1 0 0 0
57 60 1 0 0 0 0
59 61 1 0 0 0 0
54 62 1 0 0 0 0
55 27 1 0 0 0 0
58 63 1 0 0 0 0
64 65 1 0 0 0 0
34 64 1 0 0 0 0
66 67 1 0 0 0 0
15 66 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 64 65
M SBL 1 1 71
M SMT 1 CH2OH
M SBV 1 71 -0.7327 -0.3960
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 66 67
M SBL 2 1 73
M SMT 2 OCH3
M SBV 2 73 -0.7011 -0.4047
S SKP 5
ID FL3FABGS0012
FORMULA C42H54O25
EXACTMASS 958.2954172780001
AVERAGEMASS 958.86316
SMILES c(O6)(c(C(C=C6c(c7)ccc(c7)OC)=O)5)cc(cc(O)5)OC(O1)C(OC(C4O)OC(COC(C)=O)C(O)C4O)C(C(OC(C(O)3)OC(C(C(O)3)O)C)C1COC(C2O)OC(C(O)C2O)CO)O
M END