Mol:FL3FACGS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.1137 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1137 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1137 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1137 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1445 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1445 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1445 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1445 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 0.7086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 0.7086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7736 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7736 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4028 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4028 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4028 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4028 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7736 0.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7736 0.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7736 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7736 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0317 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0317 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6729 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6729 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3141 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3141 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3141 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3141 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6729 1.8191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6729 1.8191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0317 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0317 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8383 0.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8383 0.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 -1.4692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 -1.4692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1430 1.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1430 1.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6729 2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6729 2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4021 0.9521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4021 0.9521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6830 0.0028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6830 0.0028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6474 0.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6474 0.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6480 0.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6480 0.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3742 1.1426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3742 1.1426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4321 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4321 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1430 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1430 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6171 0.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6171 0.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8050 -0.1175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8050 -0.1175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8423 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8423 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4076 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4076 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3204 -1.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3204 -1.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2268 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2268 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6613 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6613 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7485 -1.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7485 -1.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0715 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0715 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9626 -1.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9626 -1.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7109 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7109 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2268 -2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2268 -2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2347 1.4338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2347 1.4338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2383 0.8585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2383 0.8585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 31 39 1 0 0 0 0 | + | 31 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 46 0.8027 -0.6710 | + | M SBV 1 46 0.8027 -0.6710 |
| − | S SKP 5 | + | S SKP 5 |
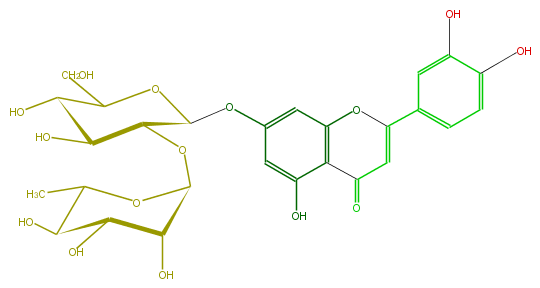
| − | ID FL3FACGS0024 | + | ID FL3FACGS0024 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(C(=O)5)(c1OC(=C5)c(c4)cc(c(c4)O)O)c(O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)c1 | + | SMILES c(C(=O)5)(c1OC(=C5)c(c4)cc(c(c4)O)O)c(O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.1137 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1137 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1445 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1445 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 0.7086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7736 -0.7443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4028 -0.3811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4028 0.3454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7736 0.7086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7736 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0317 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6729 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3141 0.7085 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3141 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6729 1.8191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0317 1.4488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8383 0.7637 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 -1.4692 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1430 1.9275 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6729 2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4021 0.9521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6830 0.0028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6474 0.4056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6480 0.4164 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3742 1.1426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4321 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1430 0.6722 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6171 0.1531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8050 -0.1175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8423 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4076 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3204 -1.5548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2268 -1.8656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6613 -0.8863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7485 -1.1969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0715 -2.2149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9626 -1.5992 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7109 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2268 -2.6906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2347 1.4338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2383 0.8585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
35 30 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
31 39 1 0 0 0 0
34 40 1 0 0 0 0
25 18 1 0 0 0 0
41 42 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^CH2OH
M SBV 1 46 0.8027 -0.6710
S SKP 5
ID FL3FACGS0024
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(C(=O)5)(c1OC(=C5)c(c4)cc(c(c4)O)O)c(O)cc(OC(C2OC(O3)C(C(O)C(O)C(C)3)O)OC(CO)C(C2O)O)c1
M END