Mol:FL3FACGS0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4336 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4336 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4336 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4336 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7982 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7982 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1629 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1629 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1629 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1629 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7982 0.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7982 0.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4726 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4726 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1080 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1080 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1080 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1080 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4726 0.1652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4726 0.1652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4726 -2.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4726 -2.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7431 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7431 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3908 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3908 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0383 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0383 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0383 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0383 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3908 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3908 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7431 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7431 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1654 0.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1654 0.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7982 -2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7982 -2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7944 1.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7944 1.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3908 2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3908 2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8654 0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8654 0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3570 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3570 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6248 -0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6248 -0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9183 -0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9183 -0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4316 0.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4316 0.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1796 0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1796 0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4338 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4338 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1496 -0.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1496 -0.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4778 0.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4778 0.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0972 -0.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0972 -0.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9007 0.4269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9007 0.4269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0972 1.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0972 1.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4778 1.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4778 1.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6743 0.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6743 0.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4338 0.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4338 0.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5351 -0.4985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5351 -0.4985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0316 -0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0316 -0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4105 -0.0307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4105 -0.0307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0416 -0.5956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0416 -0.5956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5412 0.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5412 0.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3546 0.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3546 0.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1329 1.3183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1329 1.3183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 30 37 1 0 0 0 0 | + | 30 37 1 0 0 0 0 |
| − | 31 38 1 0 0 0 0 | + | 31 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 34 20 1 0 0 0 0 | + | 34 20 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 27 41 1 0 0 0 0 | + | 27 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 41 42 43 | + | M SAL 1 3 41 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^COOH | + | M SMT 1 ^COOH |
| − | M SBV 1 47 0.3616 -0.4411 | + | M SBV 1 47 0.3616 -0.4411 |
| − | S SKP 5 | + | S SKP 5 |
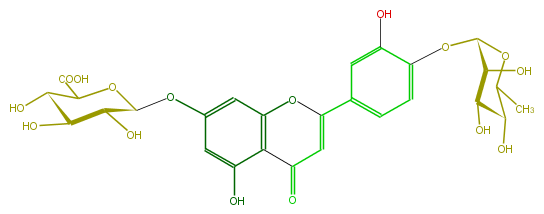
| − | ID FL3FACGS0036 | + | ID FL3FACGS0036 |
| − | FORMULA C27H28O16 | + | FORMULA C27H28O16 |
| − | EXACTMASS 608.137734848 | + | EXACTMASS 608.137734848 |
| − | AVERAGEMASS 608.50162 | + | AVERAGEMASS 608.50162 |
| − | SMILES O(c13)C(c(c4)cc(O)c(OC(C5O)OC(C(O)C5O)C)c4)=CC(=O)c(c(cc(c3)OC(O2)C(C(C(O)C2C(O)=O)O)O)O)1 | + | SMILES O(c13)C(c(c4)cc(O)c(OC(C5O)OC(C(O)C5O)C)c4)=CC(=O)c(c(cc(c3)OC(O2)C(C(C(O)C2C(O)=O)O)O)O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.4336 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4336 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7982 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1629 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1629 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7982 0.1652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4726 -1.3022 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1080 -0.9354 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1080 -0.2016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4726 0.1652 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4726 -2.0174 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7431 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3908 -0.2088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0383 0.1651 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0383 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3908 1.2867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7431 0.9128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1654 0.2209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7982 -2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7944 1.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3908 2.0344 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8654 0.3042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3570 -0.3669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6248 -0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9183 -0.0747 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4316 0.4389 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1796 0.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4338 -0.0240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1496 -0.4056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4778 0.0967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0972 -0.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9007 0.4269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0972 1.1188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4778 1.4765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6743 0.7888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4338 0.7888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5351 -0.4985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0316 -0.9026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.4105 -0.0307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0416 -0.5956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5412 0.6113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3546 0.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1329 1.3183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
35 36 1 0 0 0 0
30 37 1 0 0 0 0
31 38 1 0 0 0 0
32 39 1 0 0 0 0
34 20 1 0 0 0 0
25 18 1 0 0 0 0
24 40 1 0 0 0 0
41 42 2 0 0 0 0
41 43 1 0 0 0 0
27 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 41 42 43
M SBL 1 1 47
M SMT 1 ^COOH
M SBV 1 47 0.3616 -0.4411
S SKP 5
ID FL3FACGS0036
FORMULA C27H28O16
EXACTMASS 608.137734848
AVERAGEMASS 608.50162
SMILES O(c13)C(c(c4)cc(O)c(OC(C5O)OC(C(O)C5O)C)c4)=CC(=O)c(c(cc(c3)OC(O2)C(C(C(O)C2C(O)=O)O)O)O)1
M END