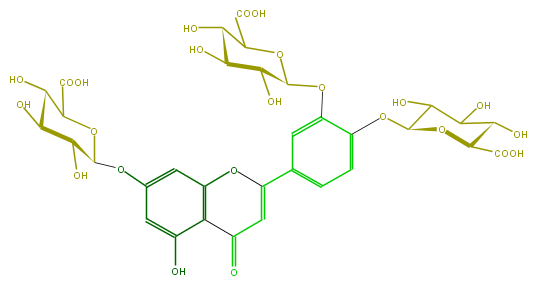
Mol:FL3FACGS0051
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0332 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0332 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0332 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0332 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3187 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3187 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6042 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6042 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6042 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6042 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3187 -1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3187 -1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8899 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8899 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1753 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1753 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1753 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1753 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8899 -1.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8899 -1.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8899 -3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8899 -3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5389 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5389 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2670 -1.5142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2670 -1.5142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9951 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9951 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9951 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9951 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2670 0.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2670 0.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5389 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5389 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6608 -1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6608 -1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3187 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3187 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7711 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7711 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4876 0.8304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4876 0.8304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6109 -0.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6109 -0.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8409 -0.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8409 -0.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3001 -0.9308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3001 -0.9308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3001 -0.1532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3001 -0.1532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0698 0.2097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0698 0.2097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1230 1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1230 1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1121 0.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1121 0.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7363 -1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7363 -1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2763 0.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2763 0.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1689 2.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1689 2.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0568 1.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0568 1.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2254 1.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2254 1.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4339 0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4339 0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2327 1.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2327 1.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6046 1.8919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6046 1.8919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8719 2.3832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8719 2.3832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7028 1.8600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7028 1.8600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0147 0.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0147 0.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9612 0.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9612 0.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3822 -0.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3822 -0.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2009 -0.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2009 -0.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9075 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9075 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4865 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4865 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6679 0.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6679 0.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2540 0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2540 0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1491 0.4851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1491 0.4851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0495 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0495 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8798 2.7336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8798 2.7336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0554 3.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0554 3.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0310 3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0310 3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0722 1.0529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0722 1.0529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4561 0.4369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4561 0.4369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0222 1.5140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0222 1.5140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5630 -0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5630 -0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1230 -1.3451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1230 -1.3451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9471 -1.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9471 -1.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 30 1 0 0 0 0 | + | 34 30 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 40 1 0 0 0 0 | + | 45 40 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 44 48 1 0 0 0 0 | + | 44 48 1 0 0 0 0 |
| − | 41 20 1 0 0 0 0 | + | 41 20 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 36 49 1 0 0 0 0 | + | 36 49 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 52 54 1 0 0 0 0 | + | 52 54 1 0 0 0 0 |
| − | 26 52 1 0 0 0 0 | + | 26 52 1 0 0 0 0 |
| − | 55 56 2 0 0 0 0 | + | 55 56 2 0 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 43 55 1 0 0 0 0 | + | 43 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 49 50 51 | + | M SAL 1 3 49 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 56 0.2752 -0.8418 | + | M SBV 1 56 0.2752 -0.8418 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 52 53 54 | + | M SAL 2 3 52 53 54 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 COOH | + | M SMT 2 COOH |
| − | M SBV 2 59 0.0024 -0.8431 | + | M SBV 2 59 0.0024 -0.8431 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 3 55 56 57 | + | M SAL 3 3 55 56 57 |
| − | M SBL 3 1 62 | + | M SBL 3 1 62 |
| − | M SMT 3 COOH | + | M SMT 3 COOH |
| − | M SBV 3 62 -0.6556 0.1469 | + | M SBV 3 62 -0.6556 0.1469 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FACGS0051 | + | ID FL3FACGS0051 |
| − | FORMULA C33H34O24 | + | FORMULA C33H34O24 |
| − | EXACTMASS 814.1440020160001 | + | EXACTMASS 814.1440020160001 |
| − | AVERAGEMASS 814.60866 | + | AVERAGEMASS 814.60866 |
| − | SMILES O(C(O6)C(O)C(C(O)C6C(O)=O)O)c(c1)cc(O)c(C2=O)c1OC(c(c3)cc(OC(C5O)OC(C(O)C5O)C(O)=O)c(OC(C(O)4)OC(C(O)=O)C(C4O)O)c3)=C2 | + | SMILES O(C(O6)C(O)C(C(O)C6C(O)=O)O)c(c1)cc(O)c(C2=O)c1OC(c(c3)cc(OC(C5O)OC(C(O)C5O)C(O)=O)c(OC(C(O)4)OC(C(O)=O)C(C4O)O)c3)=C2 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-3.0332 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0332 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3187 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6042 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6042 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3187 -1.0937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8899 -2.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1753 -2.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1753 -1.5062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8899 -1.0937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8899 -3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5389 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2670 -1.5142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9951 -1.0939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9951 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2670 0.1675 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5389 -0.2530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6608 -1.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3187 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7711 0.2075 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4876 0.8304 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6109 -0.0628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8409 -0.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3001 -0.9308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3001 -0.1532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0698 0.2097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1230 1.1153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1121 0.5061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7363 -1.2226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2763 0.9323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1689 2.3832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0568 1.4886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2254 1.3606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4339 0.9895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2327 1.7404 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6046 1.8919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8719 2.3832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7028 1.8600 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0147 0.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9612 0.4567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3822 -0.1222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2009 -0.1086 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9075 -0.5309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4865 0.0480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6679 0.0345 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2540 0.5744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1491 0.4851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.0495 -0.2117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8798 2.7336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0554 3.2239 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0310 3.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0722 1.0529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4561 0.4369 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.0222 1.5140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5630 -0.6778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.1230 -1.3451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9471 -1.2937 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
16 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 30 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 40 1 0 0 0 0
40 46 1 0 0 0 0
45 47 1 0 0 0 0
44 48 1 0 0 0 0
41 20 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
36 49 1 0 0 0 0
52 53 2 0 0 0 0
52 54 1 0 0 0 0
26 52 1 0 0 0 0
55 56 2 0 0 0 0
55 57 1 0 0 0 0
43 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 49 50 51
M SBL 1 1 56
M SMT 1 ^ COOH
M SBV 1 56 0.2752 -0.8418
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 52 53 54
M SBL 2 1 59
M SMT 2 COOH
M SBV 2 59 0.0024 -0.8431
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 3 55 56 57
M SBL 3 1 62
M SMT 3 COOH
M SBV 3 62 -0.6556 0.1469
S SKP 5
ID FL3FACGS0051
FORMULA C33H34O24
EXACTMASS 814.1440020160001
AVERAGEMASS 814.60866
SMILES O(C(O6)C(O)C(C(O)C6C(O)=O)O)c(c1)cc(O)c(C2=O)c1OC(c(c3)cc(OC(C5O)OC(C(O)C5O)C(O)=O)c(OC(C(O)4)OC(C(O)=O)C(C4O)O)c3)=C2
M END