Mol:FL3FACGS0078
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7828 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7828 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7828 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7828 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0683 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0683 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6462 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6462 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6462 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6462 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0683 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0683 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3606 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3606 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0750 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0750 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0750 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0750 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3606 0.7728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3606 0.7728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7895 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7895 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5040 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5040 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2184 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2184 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2184 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2184 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5040 2.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5040 2.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7895 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7895 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3606 -1.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3606 -1.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5024 0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5024 0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0683 -1.7350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0683 -1.7350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8413 1.9575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8413 1.9575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9241 0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9241 0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2664 1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2664 1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8538 0.5191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8538 0.5191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0554 0.7282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0554 0.7282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2619 0.5014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2619 0.5014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6745 1.2161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6745 1.2161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4729 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4729 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9080 1.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9080 1.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5680 1.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5680 1.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9241 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9241 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5952 0.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5952 0.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4527 0.1256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4527 0.1256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6989 -1.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6989 -1.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4496 -1.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4496 -1.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9786 -1.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9786 -1.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7564 -0.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7564 -0.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0058 -0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0058 -0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4768 -1.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4768 -1.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1011 -0.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1011 -0.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2470 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2470 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8399 -2.0103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8399 -2.0103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8728 -1.2644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8728 -1.2644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2718 -0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2718 -0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5476 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5476 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 7 17 2 0 0 0 0 | + | 7 17 2 0 0 0 0 |
| − | 18 1 1 0 0 0 0 | + | 18 1 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 39 43 2 0 0 0 0 | + | 39 43 2 0 0 0 0 |
| − | 39 44 1 0 0 0 0 | + | 39 44 1 0 0 0 0 |
| − | 36 21 1 0 0 0 0 | + | 36 21 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
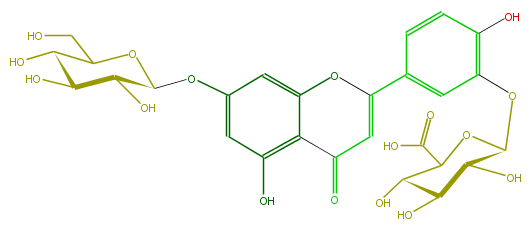
| − | ID FL3FACGS0078 | + | ID FL3FACGS0078 |
| − | FORMULA C27H28O17 | + | FORMULA C27H28O17 |
| − | EXACTMASS 624.1326494699999 | + | EXACTMASS 624.1326494699999 |
| − | AVERAGEMASS 624.50102 | + | AVERAGEMASS 624.50102 |
| − | SMILES c(c(O)4)(C(=O)1)c(cc(OC(C(O)5)OC(CO)C(O)C5O)c4)OC(c(c2)cc(OC(C3O)OC(C(O)C3O)C(O)=O)c(c2)O)=C1 | + | SMILES c(c(O)4)(C(=O)1)c(cc(OC(C(O)5)OC(CO)C(O)C5O)c4)OC(c(c2)cc(OC(C3O)OC(C(O)C3O)C(O)=O)c(c2)O)=C1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.7828 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7828 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0683 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6462 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6462 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0683 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3606 -0.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0750 -0.4647 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0750 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3606 0.7728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7895 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5040 0.3603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2184 0.7728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2184 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5040 2.0103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7895 1.5978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3606 -1.7021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5024 0.7073 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0683 -1.7350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8413 1.9575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9241 0.3653 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2664 1.2337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8538 0.5191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0554 0.7282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2619 0.5014 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6745 1.2161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4729 1.0070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9080 1.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5680 1.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9241 1.0575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5952 0.7177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4527 0.1256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6989 -1.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4496 -1.6582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9786 -1.0248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7564 -0.7493 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0058 -0.4065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4768 -1.0399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1011 -0.6374 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2470 -1.7674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8399 -2.0103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8728 -1.2644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2718 -0.0002 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5476 -0.6374 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
7 17 2 0 0 0 0
18 1 1 0 0 0 0
3 19 1 0 0 0 0
14 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 18 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
39 43 2 0 0 0 0
39 44 1 0 0 0 0
36 21 1 0 0 0 0
S SKP 5
ID FL3FACGS0078
FORMULA C27H28O17
EXACTMASS 624.1326494699999
AVERAGEMASS 624.50102
SMILES c(c(O)4)(C(=O)1)c(cc(OC(C(O)5)OC(CO)C(O)C5O)c4)OC(c(c2)cc(OC(C3O)OC(C(O)C3O)C(O)=O)c(c2)O)=C1
M END