Mol:FL3FADGS0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 39 42 0 0 0 0 0 0 0 0999 V2000 | + | 39 42 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.4673 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4673 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4673 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4673 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3694 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3694 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3694 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3694 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9183 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9183 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8205 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8205 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2715 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2715 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2715 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2715 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8205 -0.1582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8205 -0.1582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9922 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9922 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7224 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7224 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1821 -0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1821 -0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6419 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6419 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6419 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6419 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1821 0.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1821 0.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7224 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7224 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6419 0.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6419 0.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0171 -0.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0171 -0.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6223 -0.3466 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.6223 -0.3466 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.1067 -1.0273 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.1067 -1.0273 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.3642 -0.7385 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.3642 -0.7385 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6477 -0.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.6477 -0.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.1683 -0.2100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1683 -0.2100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8179 -0.5529 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.8179 -0.5529 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.3361 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3361 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9105 -1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9105 -1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9387 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9387 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2451 0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2451 0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9183 -1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9183 -1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2451 0.8753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2451 0.8753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7571 1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7571 1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2759 0.8714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2759 0.8714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7468 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7468 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2321 0.8631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2321 0.8631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7571 1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7571 1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7468 1.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7468 1.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4120 1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4120 1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8005 2.1045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8005 2.1045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 15 1 0 0 0 0 | + | 18 15 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 29 31 1 0 0 0 0 | + | 29 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 32 36 2 0 0 0 0 | + | 32 36 2 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 16 38 1 0 0 0 0 | + | 16 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 38 39 | + | M SAL 2 2 38 39 |
| − | M SBL 2 1 41 | + | M SBL 2 1 41 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 41 3.412 1.1831 | + | M SVB 2 41 3.412 1.1831 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 34 35 37 | + | M SAL 1 3 34 35 37 |
| − | M SBL 1 1 37 | + | M SBL 1 1 37 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SVB 1 37 -3.7468 1.1433 | + | M SVB 1 37 -3.7468 1.1433 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FADGS0019 | + | ID FL3FADGS0019 |
| − | KNApSAcK_ID C00004353 | + | KNApSAcK_ID C00004353 |
| − | NAME Luteolin 3'-methyl ether 7-malonylglucoside | + | NAME Luteolin 3'-methyl ether 7-malonylglucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C25H24O14 | + | FORMULA C25H24O14 |
| − | EXACTMASS 548.116605476 | + | EXACTMASS 548.116605476 |
| − | AVERAGEMASS 548.44966 | + | AVERAGEMASS 548.44966 |
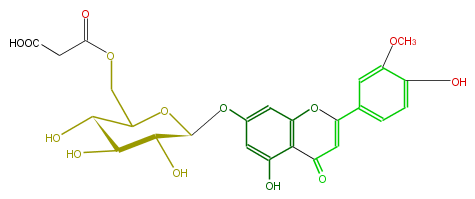
| − | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c3)cc(c(c(O)3)1)OC(c(c2)cc(c(c2)O)OC)=CC1=O | + | SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c3)cc(c(c(O)3)1)OC(c(c2)cc(c(c2)O)OC)=CC1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
39 42 0 0 0 0 0 0 0 0999 V2000
0.4673 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4673 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9183 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3694 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3694 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9183 -0.1582 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8205 -1.1999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2715 -0.9395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2715 -0.4186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8205 -0.1582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9922 -1.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7224 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1821 -0.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6419 -0.1583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6419 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1821 0.6379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7224 0.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6419 0.3725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0171 -0.1588 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6223 -0.3466 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.1067 -1.0273 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.3642 -0.7385 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6477 -0.7308 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.1683 -0.2100 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8179 -0.5529 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.3361 -0.7587 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9105 -1.0664 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9387 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2451 0.1870 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9183 -1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2451 0.8753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7571 1.1709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2759 0.8714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7468 1.1433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2321 0.8631 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7571 1.7197 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7468 1.6543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4120 1.1831 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8005 2.1045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 15 1 0 0 0 0
1 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 19 1 0 0 0 0
25 29 1 0 0 0 0
3 30 1 0 0 0 0
29 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
32 36 2 0 0 0 0
34 37 1 0 0 0 0
16 38 1 0 0 0 0
38 39 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 38 39
M SBL 2 1 41
M SMT 2 OCH3
M SVB 2 41 3.412 1.1831
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 34 35 37
M SBL 1 1 37
M SMT 1 COOH
M SVB 1 37 -3.7468 1.1433
S SKP 8
ID FL3FADGS0019
KNApSAcK_ID C00004353
NAME Luteolin 3'-methyl ether 7-malonylglucoside
CAS_RN -
FORMULA C25H24O14
EXACTMASS 548.116605476
AVERAGEMASS 548.44966
SMILES O=C(CC(O)=O)OCC([C@H](O)4)O[C@H]([C@H]([C@H]4O)O)Oc(c3)cc(c(c(O)3)1)OC(c(c2)cc(c(c2)O)OC)=CC1=O
M END