Mol:FL3FADGS0024
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.8698 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8698 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8698 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8698 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5842 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5842 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2987 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2987 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2987 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2987 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5842 1.9933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5842 1.9933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1553 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1553 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5592 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5592 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2736 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2736 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2736 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2736 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5592 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5592 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1553 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1553 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9881 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9881 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7026 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7026 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7026 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7026 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9881 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9881 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5592 -1.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5592 -1.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4171 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4171 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9881 -1.5801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9881 -1.5801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0043 1.9824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0043 1.9824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4914 -1.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4914 -1.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6945 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6945 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2780 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2780 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5568 -0.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5568 -0.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3536 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3536 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7702 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7702 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8711 -1.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8711 -1.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3503 -1.9278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3503 -1.9278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3535 -0.8370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3535 -0.8370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9623 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9623 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1755 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1755 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9136 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9136 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3605 0.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3605 0.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1474 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1474 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4094 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4094 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1564 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1564 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2927 -0.0805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2927 -0.0805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6638 1.9528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6638 1.9528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5007 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5007 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5007 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5007 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9892 -1.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9892 -1.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0152 -0.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0152 -0.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9892 -0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9892 -0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3320 -1.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3320 -1.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8732 0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8732 0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5988 0.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5988 0.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 34 37 1 0 0 0 0 | + | 34 37 1 0 0 0 0 |
| − | 30 38 1 0 0 0 0 | + | 30 38 1 0 0 0 0 |
| − | 31 20 1 0 0 0 0 | + | 31 20 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 4 45 1 0 0 0 0 | + | 4 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 39 40 41 | + | M SAL 1 3 39 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^ COOH | + | M SMT 1 ^ COOH |
| − | M SBV 1 45 0.7305 0.0000 | + | M SBV 1 45 0.7305 0.0000 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 42 43 44 | + | M SAL 2 3 42 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 COOH | + | M SMT 2 COOH |
| − | M SBV 2 48 -0.6546 0.6134 | + | M SBV 2 48 -0.6546 0.6134 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 45 46 | + | M SAL 3 2 45 46 |
| − | M SBL 3 1 50 | + | M SBL 3 1 50 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 50 -0.5745 0.3317 | + | M SBV 3 50 -0.5745 0.3317 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FADGS0024 | + | ID FL3FADGS0024 |
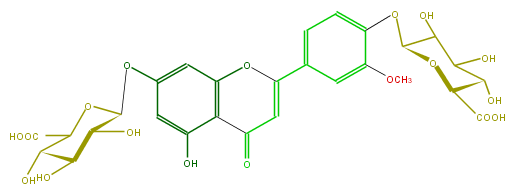
| − | FORMULA C28H28O18 | + | FORMULA C28H28O18 |
| − | EXACTMASS 652.1275640920001 | + | EXACTMASS 652.1275640920001 |
| − | AVERAGEMASS 652.51112 | + | AVERAGEMASS 652.51112 |
| − | SMILES O(C=1c(c4)ccc(OC(C5O)OC(C(O)=O)C(C5O)O)c4OC)c(c2)c(c(O)cc2OC(C(O)3)OC(C(O)=O)C(O)C3O)C(C1)=O | + | SMILES O(C=1c(c4)ccc(OC(C5O)OC(C(O)=O)C(C5O)O)c4OC)c(c2)c(c(O)cc2OC(C(O)3)OC(C(O)=O)C(O)C3O)C(C1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
0.8698 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8698 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5842 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2987 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2987 1.5808 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5842 1.9933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1553 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5592 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2736 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2736 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5592 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1553 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9881 0.7558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7026 0.3433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7026 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9881 -0.8942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5592 -1.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4171 0.7558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9881 -1.5801 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0043 1.9824 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.4914 -1.3356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6945 -1.5492 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2780 -0.8370 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5568 -0.4365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3536 -0.2229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7702 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8711 -1.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3503 -1.9278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3535 -0.8370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9623 1.4357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1755 1.1875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9136 0.8188 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3605 0.1254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1474 0.3734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4094 0.7423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1564 0.9425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2927 -0.0805 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6638 1.9528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5007 -0.9351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5007 -0.4316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9892 -1.0659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0152 -0.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9892 -0.7147 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3320 -1.2182 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8732 0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5988 0.8430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
16 19 1 0 0 0 0
5 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
35 36 1 0 0 0 0
34 37 1 0 0 0 0
30 38 1 0 0 0 0
31 20 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
26 39 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
33 42 1 0 0 0 0
45 46 1 0 0 0 0
4 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 39 40 41
M SBL 1 1 45
M SMT 1 ^ COOH
M SBV 1 45 0.7305 0.0000
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 42 43 44
M SBL 2 1 48
M SMT 2 COOH
M SBV 2 48 -0.6546 0.6134
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 45 46
M SBL 3 1 50
M SMT 3 OCH3
M SBV 3 50 -0.5745 0.3317
S SKP 5
ID FL3FADGS0024
FORMULA C28H28O18
EXACTMASS 652.1275640920001
AVERAGEMASS 652.51112
SMILES O(C=1c(c4)ccc(OC(C5O)OC(C(O)=O)C(C5O)O)c4OC)c(c2)c(c(O)cc2OC(C(O)3)OC(C(O)=O)C(O)C3O)C(C1)=O
M END