Mol:FL3FAGCS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0665 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0665 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0665 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0665 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5102 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5102 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9539 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9539 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9539 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9539 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5102 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5102 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3976 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3976 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1587 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1587 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1587 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1587 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3976 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3976 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3976 -2.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3976 -2.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6226 -0.7033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6226 -0.7033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2648 1.9813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2648 1.9813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8704 1.5225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8704 1.5225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6135 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6135 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6066 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6066 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1433 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1433 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4483 1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4483 1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6394 2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6394 2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9052 2.2377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9052 2.2377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2489 0.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2489 0.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7492 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7492 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 -1.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 -1.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9986 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9986 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9986 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9986 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 0.3597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 0.3597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7492 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7492 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6226 0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6226 0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5102 -2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5102 -2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3739 1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3739 1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6226 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6226 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0132 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0132 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2987 1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2987 1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 14 15 1 1 0 0 0 | + | 14 15 1 1 0 0 0 |
| − | 16 15 1 1 0 0 0 | + | 16 15 1 1 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 6 16 1 0 0 0 0 | + | 6 16 1 0 0 0 0 |
| − | 9 22 1 0 0 0 0 | + | 9 22 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 18 32 1 0 0 0 0 | + | 18 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 35 -7.0310 5.5381 | + | M SBV 1 35 -7.0310 5.5381 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FAGCS0002 | + | ID FL3FAGCS0002 |
| − | KNApSAcK_ID C00006108 | + | KNApSAcK_ID C00006108 |
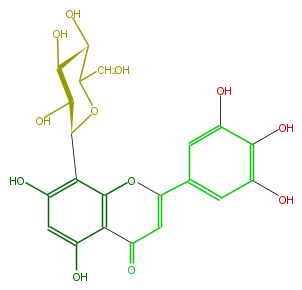
| − | NAME 5,7,3',4',5'-Pentahydroxyflavone 8-C-glucopyranoside | + | NAME 5,7,3',4',5'-Pentahydroxyflavone 8-C-glucopyranoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C21H20O12 | + | FORMULA C21H20O12 |
| − | EXACTMASS 464.095476104 | + | EXACTMASS 464.095476104 |
| − | AVERAGEMASS 464.37629999999996 | + | AVERAGEMASS 464.37629999999996 |
| − | SMILES c(c(O)1)(C(=O)3)c(OC(c(c4)cc(O)c(O)c(O)4)=C3)c(C(C(O)2)OC(CO)C(O)C2O)c(O)c1 | + | SMILES c(c(O)1)(C(=O)3)c(OC(c(c4)cc(O)c(O)c(O)4)=C3)c(C(C(O)2)OC(CO)C(O)C2O)c(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-2.0665 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0665 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5102 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9539 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9539 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5102 -0.7032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3976 -1.9879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1587 -1.6667 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1587 -1.0244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3976 -0.7032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3976 -2.4888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6226 -0.7033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2648 1.9813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8704 1.5225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6135 0.8618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6066 0.2244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1433 0.6876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4483 1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6394 2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9052 2.2377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2489 0.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7492 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 -1.0831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9986 -0.7224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9986 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 0.3597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7492 -0.0010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6226 0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5102 -2.6301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3739 1.0802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6226 -1.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0132 1.5168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2987 1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 1 1 0 0 0
14 15 1 1 0 0 0
16 15 1 1 0 0 0
16 17 1 0 0 0 0
17 18 1 0 0 0 0
18 13 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
15 21 1 0 0 0 0
6 16 1 0 0 0 0
9 22 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
25 28 1 0 0 0 0
3 29 1 0 0 0 0
26 30 1 0 0 0 0
24 31 1 0 0 0 0
18 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 CH2OH
M SBV 1 35 -7.0310 5.5381
S SKP 8
ID FL3FAGCS0002
KNApSAcK_ID C00006108
NAME 5,7,3',4',5'-Pentahydroxyflavone 8-C-glucopyranoside
CAS_RN -
FORMULA C21H20O12
EXACTMASS 464.095476104
AVERAGEMASS 464.37629999999996
SMILES c(c(O)1)(C(=O)3)c(OC(c(c4)cc(O)c(O)c(O)4)=C3)c(C(C(O)2)OC(CO)C(O)C2O)c(O)c1
M END