Mol:FL3FCACS0025
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.6187 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6187 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6187 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6187 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1750 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1750 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7313 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7313 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7313 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7313 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1750 2.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1750 2.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2876 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2876 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8439 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8439 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8439 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8439 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2876 2.2609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2876 2.2609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2876 0.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2876 0.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1750 0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1750 0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5237 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5237 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1099 2.0254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1099 2.0254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6962 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6962 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6962 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6962 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1099 3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1099 3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5237 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5237 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4078 3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4078 3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9347 1.0528 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.9347 1.0528 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -1.4191 0.4753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.4191 0.4753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9034 0.7847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9034 0.7847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2022 0.7022 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2022 0.7022 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6972 1.1559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6972 1.1559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2541 0.8878 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.2541 0.8878 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -2.4465 0.7573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4465 0.7573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0614 0.3928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0614 0.3928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5941 0.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5941 0.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2372 -0.8170 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2372 -0.8170 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.7904 -0.4976 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7904 -0.4976 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.6149 -1.1119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6149 -1.1119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7904 -1.7299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7904 -1.7299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2372 -2.0493 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.2372 -2.0493 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4127 -1.4350 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.4127 -1.4350 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.2372 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2372 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1105 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1105 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5374 -3.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5374 -3.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9690 -2.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9690 -2.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4496 -2.8769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4496 -2.8769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0991 -3.0510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0991 -3.0510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0991 -3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0991 -3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5436 -2.6065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5436 -2.6065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0469 -2.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0469 -2.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5735 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5735 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5735 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5735 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0881 -1.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0881 -1.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6027 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6027 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6027 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6027 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0881 -2.8901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0881 -2.8901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1171 -1.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1171 -1.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2614 2.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2614 2.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2387 3.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2387 3.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8309 -1.1408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8309 -1.1408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1164 -1.5533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1164 -1.5533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.4078 -2.2856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.4078 -2.2856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.3420 -1.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.3420 -1.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3217 1.4067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3217 1.4067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4649 2.2191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4649 2.2191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 2 23 1 0 0 0 0 | + | 2 23 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 30 28 1 0 0 0 0 | + | 30 28 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 1 51 1 0 0 0 0 | + | 1 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 46 53 1 0 0 0 0 | + | 46 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 48 55 1 0 0 0 0 | + | 48 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 25 57 1 0 0 0 0 | + | 25 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 57 58 | + | M SAL 4 2 57 58 |
| − | M SBL 4 1 62 | + | M SBL 4 1 62 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 62 -1.6071 1.2741 | + | M SVB 4 62 -1.6071 1.2741 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 60 | + | M SBL 3 1 60 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 60 -5.4078 -2.2856 | + | M SVB 3 60 -5.4078 -2.2856 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 58 -3.8309 -1.1408 | + | M SVB 2 58 -3.8309 -1.1408 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 56 0.2614 2.5584 | + | M SVB 1 56 0.2614 2.5584 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FCACS0025 | + | ID FL3FCACS0025 |
| − | KNApSAcK_ID C00006337 | + | KNApSAcK_ID C00006337 |
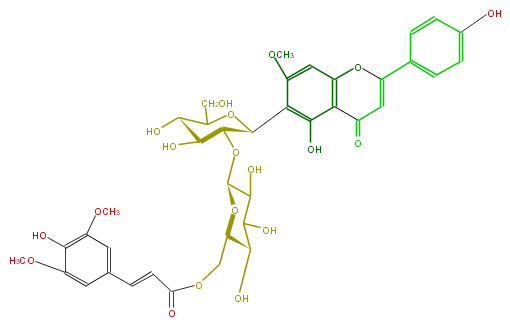
| − | NAME Spinosin 6'''-(E)-sinapoyl ester;Swertisin 6'''-O-sinapoyl 2''-O-glucoside | + | NAME Spinosin 6'''-(E)-sinapoyl ester;Swertisin 6'''-O-sinapoyl 2''-O-glucoside |
| − | CAS_RN 77690-91-6 | + | CAS_RN 77690-91-6 |
| − | FORMULA C39H42O19 | + | FORMULA C39H42O19 |
| − | EXACTMASS 814.2320291619999 | + | EXACTMASS 814.2320291619999 |
| − | AVERAGEMASS 814.73938 | + | AVERAGEMASS 814.73938 |
| − | SMILES OC(C(O)2)[C@@H](O)[C@H](O[C@H]2O[C@H]([C@@H]3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)[C@H]([C@@H](O)C(CO)O3)O)COC(=O)C=Cc(c1)cc(c(c(OC)1)O)OC | + | SMILES OC(C(O)2)[C@@H](O)[C@H](O[C@H]2O[C@H]([C@@H]3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)[C@H]([C@@H](O)C(CO)O3)O)COC(=O)C=Cc(c1)cc(c(c(OC)1)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
0.6187 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6187 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1750 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7313 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7313 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1750 2.2609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2876 0.9761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8439 1.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8439 1.9397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2876 2.2609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2876 0.4753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1750 0.3340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5237 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1099 2.0254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6962 2.3639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6962 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1099 3.3792 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5237 3.0408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4078 3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9347 1.0528 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-1.4191 0.4753 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9034 0.7847 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2022 0.7022 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6972 1.1559 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2541 0.8878 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-2.4465 0.7573 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0614 0.3928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5941 0.2488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2372 -0.8170 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.7904 -0.4976 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.6149 -1.1119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7904 -1.7299 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2372 -2.0493 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4127 -1.4350 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.2372 -0.1392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1105 -1.5752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5374 -3.1697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9690 -2.3964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4496 -2.8769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0991 -3.0510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0991 -3.5278 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5436 -2.6065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0469 -2.8971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5735 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5735 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0881 -1.7018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6027 -1.9989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6027 -2.5930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0881 -2.8901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1171 -1.7019 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2614 2.5584 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2387 3.4244 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8309 -1.1408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1164 -1.5533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.4078 -2.2856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.3420 -1.9289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3217 1.4067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4649 2.2191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
2 23 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
32 31 1 1 0 0 0
30 28 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
47 50 1 0 0 0 0
1 51 1 0 0 0 0
51 52 1 0 0 0 0
46 53 1 0 0 0 0
53 54 1 0 0 0 0
48 55 1 0 0 0 0
55 56 1 0 0 0 0
25 57 1 0 0 0 0
57 58 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 57 58
M SBL 4 1 62
M SMT 4 CH2OH
M SVB 4 62 -1.6071 1.2741
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 60
M SMT 3 OCH3
M SVB 3 60 -5.4078 -2.2856
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 58
M SMT 2 OCH3
M SVB 2 58 -3.8309 -1.1408
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 56
M SMT 1 OCH3
M SVB 1 56 0.2614 2.5584
S SKP 8
ID FL3FCACS0025
KNApSAcK_ID C00006337
NAME Spinosin 6'''-(E)-sinapoyl ester;Swertisin 6'''-O-sinapoyl 2''-O-glucoside
CAS_RN 77690-91-6
FORMULA C39H42O19
EXACTMASS 814.2320291619999
AVERAGEMASS 814.73938
SMILES OC(C(O)2)[C@@H](O)[C@H](O[C@H]2O[C@H]([C@@H]3c(c6O)c(cc(c65)OC(=CC5=O)c(c4)ccc(c4)O)OC)[C@H]([C@@H](O)C(CO)O3)O)COC(=O)C=Cc(c1)cc(c(c(OC)1)O)OC
M END