Mol:FL3FCADS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3493 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3493 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3493 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3493 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6488 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6488 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9482 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9482 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9482 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9482 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6488 -0.3807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6488 -0.3807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2477 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2477 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5472 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5472 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5472 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5472 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2477 -0.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2477 -0.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2477 -2.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2477 -2.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5059 2.1005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5059 2.1005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4201 1.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4201 1.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4006 0.9260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4006 0.9260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6671 0.1687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6671 0.1687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9193 0.5173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9193 0.5173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0313 1.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0313 1.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6936 2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6936 2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2230 2.4841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2230 2.4841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1545 0.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1545 0.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6488 -2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6488 -2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2311 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2311 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9693 -0.7810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9693 -0.7810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7075 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7075 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7075 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7075 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9693 0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9693 0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2311 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2311 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4453 0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4453 0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2942 -1.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2942 -1.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3772 -0.4556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3772 -0.4556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8185 0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8185 0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2484 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2484 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2674 0.0154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2674 0.0154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8514 -0.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8514 -0.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9626 -1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9626 -1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8568 -1.1751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8568 -1.1751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5923 0.1222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5923 0.1222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6650 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6650 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2458 1.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2458 1.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5780 0.9503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5780 0.9503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9628 -0.4817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9628 -0.4817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5923 0.6090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5923 0.6090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 12 13 1 1 0 0 0 | + | 12 13 1 1 0 0 0 |
| − | 13 14 1 1 0 0 0 | + | 13 14 1 1 0 0 0 |
| − | 15 14 1 1 0 0 0 | + | 15 14 1 1 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 13 19 1 0 0 0 0 | + | 13 19 1 0 0 0 0 |
| − | 14 20 1 0 0 0 0 | + | 14 20 1 0 0 0 0 |
| − | 6 15 1 0 0 0 0 | + | 6 15 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 9 1 0 0 0 0 | + | 22 9 1 0 0 0 0 |
| − | 25 28 1 0 0 0 0 | + | 25 28 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 17 39 1 0 0 0 0 | + | 17 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 44 -0.7855 -0.4136 | + | M SBV 1 44 -0.7855 -0.4136 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 ^ OCH3 | + | M SMT 2 ^ OCH3 |
| − | M SBV 2 46 0.6134 -0.3035 | + | M SBV 2 46 0.6134 -0.3035 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCADS0006 | + | ID FL3FCADS0006 |
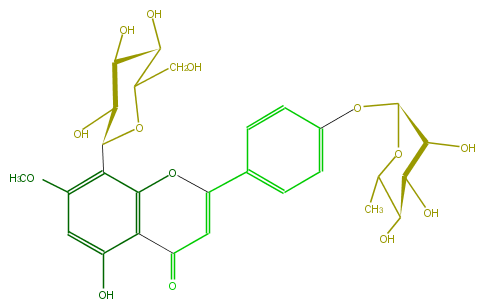
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES C(=O)(c34)C=C(Oc3c(C(C(O)5)OC(CO)C(O)C5O)c(cc(O)4)OC)c(c2)ccc(c2)OC(O1)C(O)C(C(O)C1C)O | + | SMILES C(=O)(c34)C=C(Oc3c(C(C(O)5)OC(CO)C(O)C5O)c(cc(O)4)OC)c(c2)ccc(c2)OC(O1)C(O)C(C(O)C1C)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.3493 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3493 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6488 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9482 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9482 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6488 -0.3807 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2477 -1.9985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5472 -1.5940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5472 -0.7852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2477 -0.3807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2477 -2.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5059 2.1005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4201 1.8184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4006 0.9260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6671 0.1687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9193 0.5173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0313 1.3326 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6936 2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2230 2.4841 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1545 0.4063 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6488 -2.8071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2311 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9693 -0.7810 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7075 -0.3549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7075 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9693 0.9238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2311 0.4975 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4453 0.9236 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2942 -1.2892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3772 -0.4556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8185 0.2130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2484 1.0009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2674 0.0154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8514 -0.4578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9626 -1.6886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8568 -1.1751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5923 0.1222 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6650 -1.0945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2458 1.7461 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5780 0.9503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9628 -0.4817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5923 0.6090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
12 13 1 1 0 0 0
13 14 1 1 0 0 0
15 14 1 1 0 0 0
15 16 1 0 0 0 0
16 17 1 0 0 0 0
17 12 1 0 0 0 0
12 18 1 0 0 0 0
13 19 1 0 0 0 0
14 20 1 0 0 0 0
6 15 1 0 0 0 0
3 21 1 0 0 0 0
22 23 2 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 22 1 0 0 0 0
22 9 1 0 0 0 0
25 28 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
28 32 1 0 0 0 0
34 38 1 0 0 0 0
39 40 1 0 0 0 0
17 39 1 0 0 0 0
41 42 1 0 0 0 0
1 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 CH2OH
M SBV 1 44 -0.7855 -0.4136
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 ^ OCH3
M SBV 2 46 0.6134 -0.3035
S SKP 5
ID FL3FCADS0006
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES C(=O)(c34)C=C(Oc3c(C(C(O)5)OC(CO)C(O)C5O)c(cc(O)4)OC)c(c2)ccc(c2)OC(O1)C(O)C(C(O)C1C)O
M END