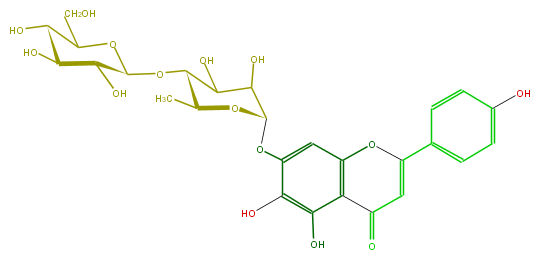
Mol:FL3FEAGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.2869 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2869 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2869 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2869 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9880 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9880 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6890 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6890 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6890 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6890 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9880 -0.6783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9880 -0.6783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3901 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3901 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0912 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0912 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0912 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0912 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3901 -0.6783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3901 -0.6783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3902 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3902 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7920 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7920 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5065 -1.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5065 -1.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2209 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2209 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2209 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2209 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5065 0.5590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5065 0.5590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7920 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7920 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2269 -0.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2269 -0.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.8541 0.5164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.8541 0.5164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0242 -3.0237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0242 -3.0237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4126 -2.2965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4126 -2.2965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9682 1.0349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9682 1.0349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6943 0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6943 0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8277 0.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8277 0.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0924 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0924 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4309 0.6335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4309 0.6335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2226 0.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2226 0.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5720 0.9585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5720 0.9585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3436 0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3436 0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5742 1.2939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5742 1.2939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2199 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2199 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9461 1.1853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9461 1.1853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0795 1.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0795 1.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3441 0.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3441 0.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6826 1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6826 1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4743 1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4743 1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.8541 1.9983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.8541 1.9983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5335 1.4843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5335 1.4843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5995 0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5995 0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3812 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3812 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8641 2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8641 2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7303 3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7303 3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 20 3 1 0 0 0 0 | + | 20 3 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 25 18 1 0 0 0 0 | + | 25 18 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 28 1 0 0 0 0 | + | 34 28 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.3898 -0.8351 | + | M SBV 1 46 0.3898 -0.8351 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0008 | + | ID FL3FEAGS0008 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c1)(ccc(C(=C5)Oc(c2C5=O)cc(OC(O4)C(C(C(C4C)OC(O3)C(O)C(O)C(C3CO)O)O)O)c(c2O)O)c1)O | + | SMILES c(c1)(ccc(C(=C5)Oc(c2C5=O)cc(OC(O4)C(C(C(C4C)OC(O3)C(O)C(O)C(C3CO)O)O)O)c(c2O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
0.2869 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2869 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9880 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6890 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6890 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9880 -0.6783 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3901 -2.2973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0912 -1.8926 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0912 -1.0832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3901 -0.6783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3902 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7920 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5065 -1.0909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2209 -0.6784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2209 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5065 0.5590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7920 0.1465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2269 -0.8036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.8541 0.5164 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0242 -3.0237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4126 -2.2965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9682 1.0349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6943 0.1472 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8277 0.1662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0924 -0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4309 0.6335 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2226 0.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5720 0.9585 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3436 0.4049 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5742 1.2939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2199 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9461 1.1853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0795 1.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3441 0.9455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6826 1.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4743 1.5628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.8541 1.9983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.5335 1.4843 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5995 0.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3812 1.3111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8641 2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7303 3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
20 3 1 0 0 0 0
2 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
25 18 1 0 0 0 0
27 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 28 1 0 0 0 0
26 40 1 0 0 0 0
41 42 1 0 0 0 0
36 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.3898 -0.8351
S SKP 5
ID FL3FEAGS0008
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c1)(ccc(C(=C5)Oc(c2C5=O)cc(OC(O4)C(C(C(C4C)OC(O3)C(O)C(O)C(C3CO)O)O)O)c(c2O)O)c1)O
M END