Mol:FL3FGCGS0005
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2918 0.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2918 0.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2512 0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2512 0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6194 1.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6194 1.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4847 1.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4847 1.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0185 1.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0185 1.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3868 1.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3868 1.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8530 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8530 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7182 2.4168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7182 2.4168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2150 2.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2150 2.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1533 2.1834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1533 2.1834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2528 1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2528 1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0803 3.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0803 3.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4556 3.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4556 3.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3183 3.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3183 3.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1945 4.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1945 4.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5698 3.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5698 3.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4324 3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4324 3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1215 0.9078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1215 0.9078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6977 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6977 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0644 5.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0644 5.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1881 0.1005 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.1881 0.1005 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.6725 -0.5801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.6725 -0.5801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.9300 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.9300 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.2136 -0.2836 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.2136 -0.2836 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.7342 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7342 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4926 -0.0352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.4926 -0.0352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.7646 -0.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7646 -0.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4763 -0.6192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4763 -0.6192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5046 -1.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5046 -1.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0877 4.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0877 4.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9436 4.5347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9436 4.5347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5529 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5529 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9678 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9678 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8600 1.6981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8600 1.6981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6349 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6349 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4130 0.3954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4130 0.3954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9433 1.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9433 1.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 2 32 1 0 0 0 0 | + | 2 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 6 34 1 0 0 0 0 | + | 6 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 26 36 1 0 0 0 0 | + | 26 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 36 37 | + | M SAL 4 2 36 37 |
| − | M SBL 4 1 39 | + | M SBL 4 1 39 |
| − | M SMT 4 CH2OH | + | M SMT 4 CH2OH |
| − | M SVB 4 39 -2.6744 0.3632 | + | M SVB 4 39 -2.6744 0.3632 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 34 35 | + | M SAL 3 2 34 35 |
| − | M SBL 3 1 37 | + | M SBL 3 1 37 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 37 0.7224 0.6492 | + | M SVB 3 37 0.7224 0.6492 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 35 | + | M SBL 2 1 35 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 35 -0.6936 -0.5572 | + | M SVB 2 35 -0.6936 -0.5572 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 33 | + | M SBL 1 1 33 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 33 2.9039 1.4694 | + | M SVB 1 33 2.9039 1.4694 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FGCGS0005 | + | ID FL3FGCGS0005 |
| − | KNApSAcK_ID C00004441 | + | KNApSAcK_ID C00004441 |
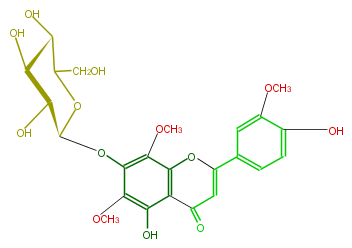
| − | NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 7-glucoside | + | NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 7-glucoside |
| − | CAS_RN 70575-23-4 | + | CAS_RN 70575-23-4 |
| − | FORMULA C24H26O13 | + | FORMULA C24H26O13 |
| − | EXACTMASS 522.137340918 | + | EXACTMASS 522.137340918 |
| − | AVERAGEMASS 522.45544 | + | AVERAGEMASS 522.45544 |
| − | SMILES C(C([C@H](O)4)O[C@H]([C@@H](O)[C@H]4O)Oc(c(OC)3)c(c(O1)c(c(O)3)C(=O)C=C1c(c2)cc(c(O)c2)OC)OC)O | + | SMILES C(C([C@H](O)4)O[C@H]([C@@H](O)[C@H]4O)Oc(c(OC)3)c(c(O1)c(c(O)3)C(=O)C=C1c(c2)cc(c(O)c2)OC)OC)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-0.2918 0.8196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2512 0.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6194 1.0423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4847 1.5454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0185 1.6803 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3868 1.3119 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8530 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7182 2.4168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2150 2.5516 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1533 2.1834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2528 1.9844 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0803 3.0545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4556 3.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3183 3.9427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1945 4.0801 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5698 3.7046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4324 3.1919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1215 0.9078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6977 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0644 5.0460 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1881 0.1005 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.6725 -0.5801 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9300 -0.2913 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.2136 -0.2836 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.7342 0.2371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4926 -0.0352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.7646 -0.2323 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4763 -0.6192 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5046 -1.0055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0877 4.0175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9436 4.5347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5529 0.0132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9678 -0.8964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8600 1.6981 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6349 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4130 0.3954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9433 1.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 19 1 0 0 0 0
16 30 1 0 0 0 0
30 31 1 0 0 0 0
2 32 1 0 0 0 0
32 33 1 0 0 0 0
6 34 1 0 0 0 0
34 35 1 0 0 0 0
26 36 1 0 0 0 0
36 37 1 0 0 0 0
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 36 37
M SBL 4 1 39
M SMT 4 CH2OH
M SVB 4 39 -2.6744 0.3632
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 34 35
M SBL 3 1 37
M SMT 3 OCH3
M SVB 3 37 0.7224 0.6492
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 35
M SMT 2 OCH3
M SVB 2 35 -0.6936 -0.5572
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 33
M SMT 1 OCH3
M SVB 1 33 2.9039 1.4694
S SKP 8
ID FL3FGCGS0005
KNApSAcK_ID C00004441
NAME 5,7,4'-Trihydroxy-6,8,3'-trimethoxyflavone 7-glucoside
CAS_RN 70575-23-4
FORMULA C24H26O13
EXACTMASS 522.137340918
AVERAGEMASS 522.45544
SMILES C(C([C@H](O)4)O[C@H]([C@@H](O)[C@H]4O)Oc(c(OC)3)c(c(O1)c(c(O)3)C(=O)C=C1c(c2)cc(c(O)c2)OC)OC)O
M END