Mol:FL4D1CGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2411 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2411 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7202 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7202 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1994 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1994 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1994 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1994 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7202 0.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7202 0.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2411 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2411 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3215 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3215 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8424 -0.5321 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8424 -0.5321 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.8424 0.0694 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.8424 0.0694 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.3215 0.3701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3215 0.3701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3215 -1.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3215 -1.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4221 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4221 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9508 0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9508 0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4796 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4796 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4796 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4796 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9508 1.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9508 1.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4221 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4221 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7743 0.3772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7743 0.3772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3106 -1.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3106 -1.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0083 1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0083 1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8168 0.2121 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -3.8168 0.2121 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -3.4727 -0.2421 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -3.4727 -0.2421 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.9772 -0.0494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.9772 -0.0494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.4991 -0.0443 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.4991 -0.0443 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -2.8465 0.3033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8465 0.3033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3527 0.1215 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -3.3527 0.1215 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -4.5320 0.2748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5320 0.2748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7976 -0.6697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7976 -0.6697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6933 -0.5260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6933 -0.5260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6498 -0.9900 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.6498 -0.9900 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.3816 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3816 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.8974 -1.3071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8974 -1.3071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4163 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.4163 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6846 -0.9900 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.6846 -0.9900 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1687 -1.1374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.1687 -1.1374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.2852 -0.6253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2852 -0.6253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2057 -0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2057 -0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1045 -0.8371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1045 -0.8371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9508 1.8951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9508 1.8951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2714 -1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2714 -1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2501 -2.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2501 -2.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5523 0.9994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5523 0.9994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5076 1.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5076 1.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 31 19 1 0 0 0 0 | + | 31 19 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 26 42 1 0 0 0 0 | + | 26 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 -3.5523 0.9994 | + | M SVB 2 46 -3.5523 0.9994 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 3.7242 -1.4826 | + | M SVB 1 44 3.7242 -1.4826 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL4D1CGS0003 | + | ID FL4D1CGS0003 |
| − | KNApSAcK_ID C00008688 | + | KNApSAcK_ID C00008688 |
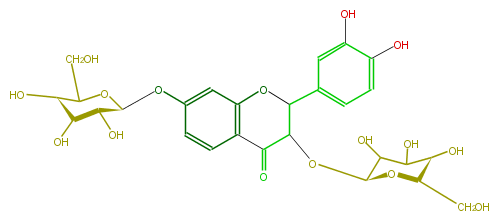
| − | NAME Fustin 3,7-diglucoside | + | NAME Fustin 3,7-diglucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C27H32O16 | + | FORMULA C27H32O16 |
| − | EXACTMASS 612.1690349759999 | + | EXACTMASS 612.1690349759999 |
| − | AVERAGEMASS 612.53338 | + | AVERAGEMASS 612.53338 |
| − | SMILES C([C@@H](O1)[C@@H](C(O)C([C@@H]1OC(C(c(c5)ccc(O)c5O)4)C(=O)c(c(O4)2)ccc(O[C@H](O3)[C@@H](O)[C@@H](O)[C@@H](O)C3CO)c2)O)O)O | + | SMILES C([C@@H](O1)[C@@H](C(O)C([C@@H]1OC(C(c(c5)ccc(O)c5O)4)C(=O)c(c(O4)2)ccc(O[C@H](O3)[C@@H](O)[C@@H](O)[C@@H](O)C3CO)c2)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.2411 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7202 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1994 -0.5321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1994 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7202 0.3701 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2411 0.0694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3215 -0.8328 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8424 -0.5321 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.8424 0.0694 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.3215 0.3701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3215 -1.3567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4221 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9508 0.0636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4796 0.3688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4796 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9508 1.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4221 0.9793 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7743 0.3772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3106 -1.1088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0083 1.2846 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8168 0.2121 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-3.4727 -0.2421 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.9772 -0.0494 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.4991 -0.0443 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-2.8465 0.3033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3527 0.1215 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-4.5320 0.2748 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7976 -0.6697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6933 -0.5260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6498 -0.9900 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.3816 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.8974 -1.3071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4163 -1.4546 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6846 -0.9900 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1687 -1.1374 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.2852 -0.6253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2057 -0.7142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1045 -0.8371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9508 1.8951 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2714 -1.9730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2501 -2.9728 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5523 0.9994 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5076 1.2950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
31 19 1 0 0 0 0
16 39 1 0 0 0 0
33 40 1 0 0 0 0
40 41 1 0 0 0 0
26 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 -3.5523 0.9994
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 3.7242 -1.4826
S SKP 8
ID FL4D1CGS0003
KNApSAcK_ID C00008688
NAME Fustin 3,7-diglucoside
CAS_RN -
FORMULA C27H32O16
EXACTMASS 612.1690349759999
AVERAGEMASS 612.53338
SMILES C([C@@H](O1)[C@@H](C(O)C([C@@H]1OC(C(c(c5)ccc(O)c5O)4)C(=O)c(c(O4)2)ccc(O[C@H](O3)[C@@H](O)[C@@H](O)[C@@H](O)C3CO)c2)O)O)O
M END