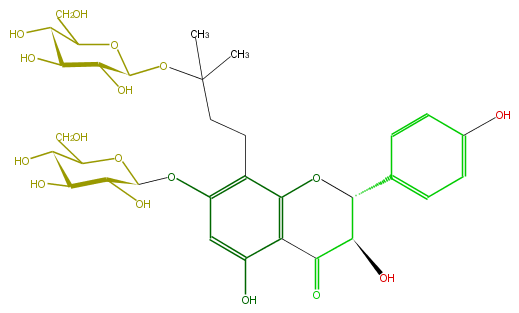
Mol:FL4DAAGI0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.7977 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7977 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0826 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0826 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6326 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6326 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6324 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6324 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0826 -0.8145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0826 -0.8145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7978 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7978 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3477 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3477 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0628 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0628 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0629 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0629 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3477 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3477 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3476 -3.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3476 -3.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8588 -0.8163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8588 -0.8163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5846 -1.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5846 -1.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3106 -0.8162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3106 -0.8162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3106 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3106 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5845 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5845 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8588 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8588 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5697 -0.8061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5697 -0.8061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7057 -2.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7057 -2.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0366 0.4410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0366 0.4410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9698 -0.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9698 -0.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6130 -1.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6130 -1.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8970 -0.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8970 -0.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2494 -0.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2494 -0.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6362 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6362 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3640 -0.5465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3640 -0.5465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5186 -0.4819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5186 -0.4819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1853 -0.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1853 -0.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2060 -1.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2060 -1.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0826 -3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0826 -3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0825 0.0104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0825 0.0104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8043 0.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8043 0.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9475 1.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9475 1.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7228 1.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7228 1.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3155 1.6791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3155 1.6791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0131 2.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0131 2.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7824 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7824 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0527 1.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0527 1.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4334 1.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4334 1.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7186 1.8472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7186 1.8472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4568 1.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4568 1.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6133 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6133 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3880 1.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3880 1.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5805 0.9382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5805 0.9382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1126 2.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1126 2.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9367 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9367 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0366 3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0366 3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9382 -0.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9382 -0.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7508 0.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7508 0.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 5 31 1 0 0 0 0 | + | 5 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 33 45 1 0 0 0 0 | + | 33 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 41 46 1 0 0 0 0 | + | 41 46 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 26 48 1 0 0 0 0 | + | 26 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 46 47 | + | M SAL 1 2 46 47 |
| − | M SBL 1 1 51 | + | M SBL 1 1 51 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 51 0.4798 -0.6190 | + | M SBV 1 51 0.4798 -0.6190 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 48 49 | + | M SAL 2 2 48 49 |
| − | M SBL 2 1 53 | + | M SBL 2 1 53 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 53 0.5742 -0.5379 | + | M SBV 2 53 0.5742 -0.5379 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL4DAAGI0003 | + | ID FL4DAAGI0003 |
| − | FORMULA C32H42O17 | + | FORMULA C32H42O17 |
| − | EXACTMASS 698.242199918 | + | EXACTMASS 698.242199918 |
| − | AVERAGEMASS 698.6656800000001 | + | AVERAGEMASS 698.6656800000001 |
| − | SMILES C(c(c5)ccc(c5)O)(O4)C(C(c(c42)c(cc(c2CCC(C)(C)OC(O3)C(O)C(C(C(CO)3)O)O)OC(O1)C(C(O)C(C(CO)1)O)O)O)=O)O | + | SMILES C(c(c5)ccc(c5)O)(O4)C(C(c(c42)c(cc(c2CCC(C)(C)OC(O3)C(O)C(C(C(CO)3)O)O)OC(O1)C(C(O)C(C(CO)1)O)O)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-0.7977 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0826 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6326 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6324 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0826 -0.8145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7978 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3477 -2.4662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0628 -2.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0629 -1.2276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3477 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3476 -3.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8588 -0.8163 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5846 -1.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3106 -0.8162 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3106 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5845 0.4409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8588 0.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5697 -0.8061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7057 -2.8450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0366 0.4410 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9698 -0.3134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6130 -1.0096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8970 -0.8672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2494 -0.9742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6362 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3640 -0.5465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5186 -0.4819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1853 -0.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2060 -1.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0826 -3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0825 0.0104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8043 0.3362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9475 1.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7228 1.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3155 1.6791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0131 2.1854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7824 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0527 1.4537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4334 1.2359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7186 1.8472 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4568 1.8505 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6133 2.0780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3880 1.5868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5805 0.9382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1126 2.0850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9367 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0366 3.2911 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9382 -0.0086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7508 0.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 18 1 0 0 0 0
2 30 1 0 0 0 0
5 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
33 35 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
39 34 1 0 0 0 0
33 45 1 0 0 0 0
46 47 1 0 0 0 0
41 46 1 0 0 0 0
48 49 1 0 0 0 0
26 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 46 47
M SBL 1 1 51
M SMT 1 ^ CH2OH
M SBV 1 51 0.4798 -0.6190
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 48 49
M SBL 2 1 53
M SMT 2 ^ CH2OH
M SBV 2 53 0.5742 -0.5379
S SKP 5
ID FL4DAAGI0003
FORMULA C32H42O17
EXACTMASS 698.242199918
AVERAGEMASS 698.6656800000001
SMILES C(c(c5)ccc(c5)O)(O4)C(C(c(c42)c(cc(c2CCC(C)(C)OC(O3)C(O)C(C(C(CO)3)O)O)OC(O1)C(C(O)C(C(CO)1)O)O)O)=O)O
M END