Mol:FL4DACGS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3954 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3954 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8745 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8745 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3536 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3536 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3536 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3536 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8745 0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8745 0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3954 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3954 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1673 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1673 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6882 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6882 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6882 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6882 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1673 0.6373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1673 0.6373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1673 -1.0895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1673 -1.0895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2679 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2679 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7966 0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7966 0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3253 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3253 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3253 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3253 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7966 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7966 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2679 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2679 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8460 0.6238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8460 0.6238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1564 -0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1564 -0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8540 1.5518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8540 1.5518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8745 -1.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8745 -1.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7966 2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7966 2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6605 -1.4196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6605 -1.4196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3143 -1.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3143 -1.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8157 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8157 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2960 -1.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2960 -1.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6842 -1.3279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6842 -1.3279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1935 -1.5107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1935 -1.5107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6605 -0.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6605 -0.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8540 -1.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8540 -1.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5300 -2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5300 -2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9573 -0.9618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9573 -0.9618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2428 -1.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2428 -1.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 35 -4.8113 3.9973 | + | M SBV 1 35 -4.8113 3.9973 |
| − | S SKP 8 | + | S SKP 8 |
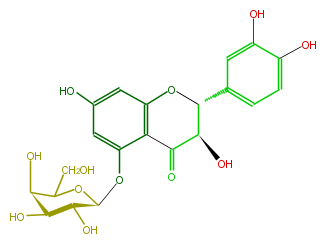
| − | ID FL4DACGS0015 | + | ID FL4DACGS0015 |
| − | KNApSAcK_ID C00008707 | + | KNApSAcK_ID C00008707 |
| − | NAME Taxifolin 5-galactoside | + | NAME Taxifolin 5-galactoside |
| − | CAS_RN 81074-97-7 | + | CAS_RN 81074-97-7 |
| − | FORMULA C21H22O12 | + | FORMULA C21H22O12 |
| − | EXACTMASS 466.111126168 | + | EXACTMASS 466.111126168 |
| − | AVERAGEMASS 466.39218 | + | AVERAGEMASS 466.39218 |
| − | SMILES C(C1Oc(c32)cc(O)cc2OC(c(c4)cc(c(O)c4)O)C(C3=O)O)(O)C(O)C(O)C(O1)CO | + | SMILES C(C1Oc(c32)cc(O)cc2OC(c(c4)cc(c(O)c4)O)C(C3=O)O)(O)C(O)C(O)C(O1)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-1.3954 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8745 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3536 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3536 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8745 0.6373 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3954 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1673 -0.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6882 -0.2649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6882 0.3366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1673 0.6373 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1673 -1.0895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2679 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7966 0.3308 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3253 0.6360 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3253 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7966 1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2679 1.2465 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8460 0.6238 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1564 -0.8416 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8540 1.5518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8745 -1.1665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7966 2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6605 -1.4196 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3143 -1.8766 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8157 -1.6827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2960 -1.6885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6842 -1.3279 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1935 -1.5107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6605 -0.6714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8540 -1.9029 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5300 -2.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9573 -0.9618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2428 -1.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
2 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 21 1 0 0 0 0
28 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 CH2OH
M SBV 1 35 -4.8113 3.9973
S SKP 8
ID FL4DACGS0015
KNApSAcK_ID C00008707
NAME Taxifolin 5-galactoside
CAS_RN 81074-97-7
FORMULA C21H22O12
EXACTMASS 466.111126168
AVERAGEMASS 466.39218
SMILES C(C1Oc(c32)cc(O)cc2OC(c(c4)cc(c(O)c4)O)C(C3=O)O)(O)C(O)C(O)C(O1)CO
M END