Mol:FL5FAAGL0090
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 48 52 0 0 0 0 0 0 0 0999 V2000 | + | 48 52 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.3914 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3914 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3914 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3914 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6769 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6769 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9624 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9624 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9624 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9624 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6769 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6769 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2479 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2479 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5334 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5334 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5334 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5334 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2479 2.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2479 2.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2479 -0.0399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2479 -0.0399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6336 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6336 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0946 1.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0946 1.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8229 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8229 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8229 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8229 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0946 3.6736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0946 3.6736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6336 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6336 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6769 -0.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6769 -0.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6023 3.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6023 3.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0537 2.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0537 2.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5924 0.2577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5924 0.2577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9010 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9010 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5147 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5147 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2577 3.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2577 3.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0052 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0052 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3916 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3916 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6486 3.9161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6486 3.9161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2054 4.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2054 4.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0745 4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0745 4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0537 3.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0537 3.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1337 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1337 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8444 -0.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8444 -0.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6188 -1.2690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6188 -1.2690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8444 -2.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8444 -2.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1337 -2.4730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1337 -2.4730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3591 -1.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3591 -1.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1317 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1317 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2068 -2.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2068 -2.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2345 -2.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2345 -2.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7269 0.3262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7269 0.3262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7269 0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7269 0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5007 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5007 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2858 0.3327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2858 0.3327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5007 -0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5007 -0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7954 -3.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7954 -3.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1206 -4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1206 -4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6961 3.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6961 3.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6961 2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6961 2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 32 21 1 0 0 0 0 | + | 32 21 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 34 45 1 0 0 0 0 | + | 34 45 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 25 47 1 0 0 0 0 | + | 25 47 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 45 46 | + | M SAL 1 2 45 46 |
| − | M SBL 1 1 50 | + | M SBL 1 1 50 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 50 -0.0490 1.1572 | + | M SBV 1 50 -0.0490 1.1572 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 47 48 | + | M SAL 2 2 47 48 |
| − | M SBL 2 1 52 | + | M SBL 2 1 52 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 52 -0.6909 -0.0769 | + | M SBV 2 52 -0.6909 -0.0769 |
| − | S SKP 5 | + | S SKP 5 |
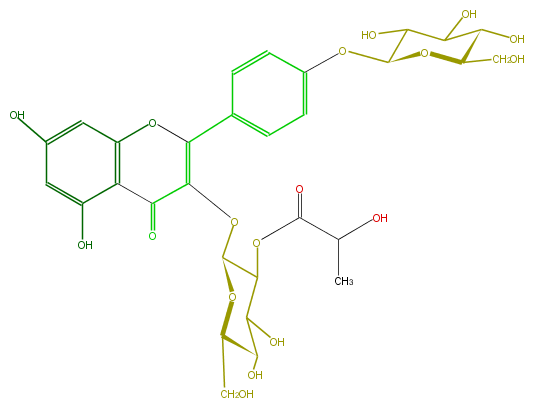
| − | ID FL5FAAGL0090 | + | ID FL5FAAGL0090 |
| − | FORMULA C30H34O18 | + | FORMULA C30H34O18 |
| − | EXACTMASS 682.174514284 | + | EXACTMASS 682.174514284 |
| − | AVERAGEMASS 682.58016 | + | AVERAGEMASS 682.58016 |
| − | SMILES C(=C3c(c4)ccc(OC(O5)C(C(O)C(O)C5CO)O)c4)(C(c(c(O3)2)c(O)cc(O)c2)=O)OC(O1)C(C(C(C(CO)1)O)O)OC(C(C)O)=O | + | SMILES C(=C3c(c4)ccc(OC(O5)C(C(O)C(O)C5CO)O)c4)(C(c(c(O3)2)c(O)cc(O)c2)=O)OC(O1)C(C(C(C(CO)1)O)O)OC(C(C)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
48 52 0 0 0 0 0 0 0 0999 V2000
-4.3914 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3914 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6769 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9624 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9624 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6769 2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2479 0.6034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5334 1.0159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5334 1.8410 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2479 2.2534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2479 -0.0399 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6336 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0946 1.9918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8229 2.4123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8229 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0946 3.6736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6336 3.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6769 -0.2214 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6023 3.7031 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0537 2.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5924 0.2577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9010 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5147 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2577 3.6715 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0052 3.4592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3916 4.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6486 3.9161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2054 4.0378 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0745 4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0537 3.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1337 -0.8903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8444 -0.4800 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6188 -1.2690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8444 -2.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1337 -2.4730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3591 -1.6840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1317 -0.1696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2068 -2.1752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2345 -2.8413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7269 0.3262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7269 0.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5007 -0.1205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2858 0.3327 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5007 -0.9406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7954 -3.2198 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1206 -4.4624 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6961 3.5361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6961 2.4765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
32 21 1 0 0 0 0
37 40 1 0 0 0 0
40 41 2 0 0 0 0
40 42 1 0 0 0 0
42 43 1 0 0 0 0
42 44 1 0 0 0 0
45 46 1 0 0 0 0
34 45 1 0 0 0 0
47 48 1 0 0 0 0
25 47 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 45 46
M SBL 1 1 50
M SMT 1 CH2OH
M SBV 1 50 -0.0490 1.1572
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 47 48
M SBL 2 1 52
M SMT 2 CH2OH
M SBV 2 52 -0.6909 -0.0769
S SKP 5
ID FL5FAAGL0090
FORMULA C30H34O18
EXACTMASS 682.174514284
AVERAGEMASS 682.58016
SMILES C(=C3c(c4)ccc(OC(O5)C(C(O)C(O)C5CO)O)c4)(C(c(c(O3)2)c(O)cc(O)c2)=O)OC(O1)C(C(C(C(CO)1)O)O)OC(C(C)O)=O
M END