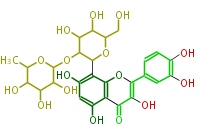
Mol:FL5FACCS0002
From Metabolomics.JP
(Difference between revisions)
| (2 intermediate revisions by one user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.9391 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9391 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2246 -0.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2246 -0.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4899 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4899 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4899 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4899 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2246 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2246 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9391 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9391 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2043 -0.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2043 -0.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9187 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9187 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9187 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9187 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2043 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2043 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5644 -0.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5644 -0.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2043 -2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2043 -2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2246 -2.6618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2246 -2.6618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7208 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7208 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4352 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4352 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1497 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1497 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1497 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1497 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4352 0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4352 0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7208 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7208 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7649 0.7792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7649 0.7792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5761 -2.0450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5761 -2.0450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8464 -0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8464 -0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5910 2.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5910 2.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3240 1.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3240 1.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1519 1.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1519 1.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4152 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4152 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6821 0.6331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6821 0.6331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8541 1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8541 1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3841 1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3841 1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2488 2.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2488 2.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7613 2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7613 2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2447 2.4115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2447 2.4115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8644 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8644 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5985 1.9030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5985 1.9030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3131 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3131 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0863 1.5221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0863 1.5221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2956 0.7238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2956 0.7238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5810 0.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5810 0.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8077 1.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8077 1.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8464 0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8464 0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6662 1.9969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6662 1.9969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1189 2.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1189 2.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6373 2.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6373 2.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 1 14 1 0 0 0 0 | + | 1 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 6 21 1 0 0 0 0 | + | 6 21 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 7 26 1 0 0 0 0 | + | 7 26 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 34 43 1 0 0 0 0 | + | 34 43 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID | + | ID FL5FACCS0002 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES C(O)C(O2)C(C(O)C(C2c(c(O)5)c(O3)c(c(c5)O)C(C(O)=C3c(c4)ccc(O)c(O)4)=O)OC(O1)C(O)C(O)C(C1C)O)O | + | SMILES C(O)C(O2)C(C(O)C(C2c(c(O)5)c(O3)c(c(c5)O)C(C(O)=C3c(c4)ccc(O)c(O)4)=O)OC(O1)C(O)C(O)C(C1C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 12:34, 30 September 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.9391 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2246 -0.4398 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4899 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4899 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2246 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9391 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2043 -0.4398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9187 -0.8523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9187 -1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2043 -2.0898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5644 -0.4795 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2043 -2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2246 -2.6618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7208 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4352 -0.8134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1497 -0.4009 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1497 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4352 0.8365 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7208 0.4240 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7649 0.7792 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5761 -2.0450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8464 -0.8032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5910 2.2225 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3240 1.8433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1519 1.0362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4152 0.2540 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6821 0.6331 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8541 1.4403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3841 1.6719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2488 2.3845 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7613 2.8293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2447 2.4115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8644 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5985 1.9030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3131 2.3156 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0863 1.5221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2956 0.7238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5810 0.3111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8077 1.1047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8464 0.6872 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6662 1.9969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1189 2.7913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6373 2.5343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
1 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
6 21 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
7 26 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
34 43 1 0 0 0 0
38 33 1 0 0 0 0
S SKP 5
ID FL5FACCS0002
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES C(O)C(O2)C(C(O)C(C2c(c(O)5)c(O3)c(c(c5)O)C(C(O)=C3c(c4)ccc(O)c(O)4)=O)OC(O1)C(O)C(O)C(C1C)O)O
M END