Mol:FL5FACGA0027
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.2126 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2126 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2126 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2126 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6563 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6563 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1000 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1000 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1000 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1000 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6563 0.9822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6563 0.9822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5437 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5437 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9874 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9874 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9874 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9874 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5437 0.9822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5437 0.9822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5437 -0.8033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5437 -0.8033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4313 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4313 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1357 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1357 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7026 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7026 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7026 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7026 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1357 1.9641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1357 1.9641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4313 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4313 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3393 -0.5316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3393 -0.5316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7061 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7061 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3185 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3185 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0639 -0.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0639 -0.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8139 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8139 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2016 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2016 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4561 -0.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4561 -0.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0210 -0.2350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0210 -0.2350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7249 0.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7249 0.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2016 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2016 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2694 1.9640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2694 1.9640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6563 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6563 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8236 0.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8236 0.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4429 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4429 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7884 -1.3102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7884 -1.3102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1605 -1.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1605 -1.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8236 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8236 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1605 -2.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1605 -2.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1357 2.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1357 2.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 16 36 1 0 0 0 0 | + | 16 36 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGA0027 | + | ID FL5FACGA0027 |
| − | KNApSAcK_ID C00005946 | + | KNApSAcK_ID C00005946 |
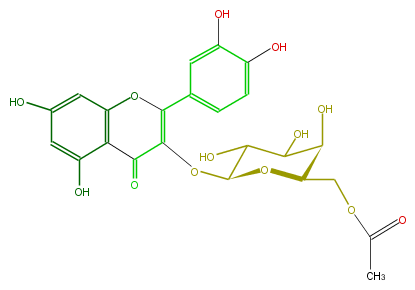
| − | NAME Quercetin 3-(6''-acetylgalactoside) | + | NAME Quercetin 3-(6''-acetylgalactoside) |
| − | CAS_RN 94799-71-0 | + | CAS_RN 94799-71-0 |
| − | FORMULA C23H22O13 | + | FORMULA C23H22O13 |
| − | EXACTMASS 506.10604078999995 | + | EXACTMASS 506.10604078999995 |
| − | AVERAGEMASS 506.41298000000006 | + | AVERAGEMASS 506.41298000000006 |
| − | SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O | + | SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-3.2126 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2126 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6563 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1000 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1000 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6563 0.9822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5437 -0.3025 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9874 0.0187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9874 0.6610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5437 0.9822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5437 -0.8033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4313 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1357 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7026 0.9821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7026 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1357 1.9641 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4313 1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3393 -0.5316 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7061 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3185 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0639 -0.4986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8139 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2016 -0.0402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4561 -0.2532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0210 -0.2350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7249 0.2124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2016 0.7230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2694 1.9640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6563 -0.9446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8236 0.8595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4429 -0.7116 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7884 -1.3102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1605 -1.8758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8236 -1.4930 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1605 -2.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1357 2.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
16 36 1 0 0 0 0
S SKP 8
ID FL5FACGA0027
KNApSAcK_ID C00005946
NAME Quercetin 3-(6''-acetylgalactoside)
CAS_RN 94799-71-0
FORMULA C23H22O13
EXACTMASS 506.10604078999995
AVERAGEMASS 506.41298000000006
SMILES c(c(C(O3)=C(C(c(c4O)c3cc(c4)O)=O)OC(O2)C(O)C(C(C(COC(C)=O)2)O)O)1)cc(c(c1)O)O
M END