Mol:FL5FACGL0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3802 -4.5467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3802 -4.5467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7597 -4.7130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7597 -4.7130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3055 -4.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3055 -4.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4718 -3.6383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4718 -3.6383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0923 -3.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0923 -3.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5465 -3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5465 -3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0175 -3.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0175 -3.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1838 -2.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1838 -2.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8043 -2.3973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8043 -2.3973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2586 -2.8515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2586 -2.8515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4661 -3.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4661 -3.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9705 -1.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9705 -1.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5076 -1.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5076 -1.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6770 -0.6818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6770 -0.6818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3094 -0.5124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3094 -0.5124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7724 -0.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7724 -0.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6028 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6028 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8342 -5.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8342 -5.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5154 -2.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5154 -2.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2472 -0.8198 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.2472 -0.8198 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.8596 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.8596 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.6050 -1.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6050 -1.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3550 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3550 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7427 -0.8198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7427 -0.8198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9972 -1.0328 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 1.9972 -1.0328 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.5201 -1.0146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5201 -1.0146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2992 -0.5096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2992 -0.5096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2276 -0.8198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2276 -0.8198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4788 0.1198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4788 0.1198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3147 -4.4248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3147 -4.4248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4046 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4046 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9383 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9383 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9241 -2.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9241 -2.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 16 31 1 0 0 0 0 | + | 16 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 32 33 | + | M SAL 1 2 32 33 |
| − | M SBL 1 1 35 | + | M SBL 1 1 35 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 35 2.9383 -1.8391 | + | M SVB 1 35 2.9383 -1.8391 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGL0001 | + | ID FL5FACGL0001 |
| − | KNApSAcK_ID C00005373 | + | KNApSAcK_ID C00005373 |
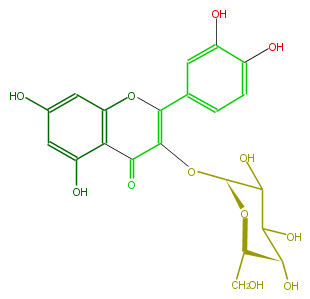
| − | NAME Isoquercitrin;Hirsutrin;Quercetin 3-beta-D-glucoside | + | NAME Isoquercitrin;Hirsutrin;Quercetin 3-beta-D-glucoside |
| − | CAS_RN 482-35-9 | + | CAS_RN 482-35-9 |
| − | FORMULA C21H20O12 | + | FORMULA C21H20O12 |
| − | EXACTMASS 464.095476104 | + | EXACTMASS 464.095476104 |
| − | AVERAGEMASS 464.37629999999996 | + | AVERAGEMASS 464.37629999999996 |
| − | SMILES O(C(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@@H](C(O)1)O[C@H](CO)[C@H](O)C1O | + | SMILES O(C(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@@H](C(O)1)O[C@H](CO)[C@H](O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-1.3802 -4.5467 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7597 -4.7130 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3055 -4.2588 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4718 -3.6383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0923 -3.4720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5465 -3.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0175 -3.1841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1838 -2.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8043 -2.3973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2586 -2.8515 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4661 -3.3136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9705 -1.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5076 -1.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6770 -0.6818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3094 -0.5124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7724 -0.9753 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6028 -1.6076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8342 -5.0008 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5154 -2.0800 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2472 -0.8198 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.8596 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.6050 -1.2781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3550 -1.4912 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7427 -0.8198 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9972 -1.0328 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.5201 -1.0146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2992 -0.5096 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2276 -0.8198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4788 0.1198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3147 -4.4248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4046 -0.8059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9383 -1.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9241 -2.8390 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
23 22 1 1 0 0 0
15 29 1 0 0 0 0
3 30 1 0 0 0 0
16 31 1 0 0 0 0
23 32 1 0 0 0 0
32 33 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 32 33
M SBL 1 1 35
M SMT 1 CH2OH
M SVB 1 35 2.9383 -1.8391
S SKP 8
ID FL5FACGL0001
KNApSAcK_ID C00005373
NAME Isoquercitrin;Hirsutrin;Quercetin 3-beta-D-glucoside
CAS_RN 482-35-9
FORMULA C21H20O12
EXACTMASS 464.095476104
AVERAGEMASS 464.37629999999996
SMILES O(C(C(=O)3)=C(Oc(c4)c3c(cc(O)4)O)c(c2)cc(c(O)c2)O)[C@@H](C(O)1)O[C@H](CO)[C@H](O)C1O
M END