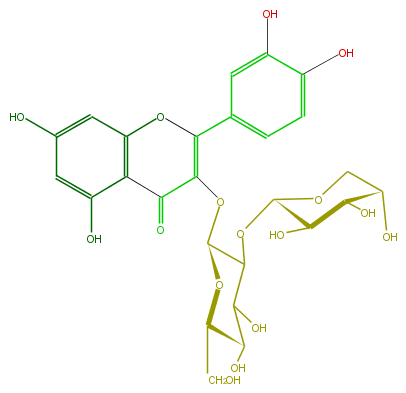
Mol:FL5FACGL0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9579 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9579 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9579 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9579 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2567 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2567 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5555 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5555 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5555 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5555 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2567 1.5990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2567 1.5990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8543 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8543 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1530 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1530 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1530 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1530 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8543 1.5990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8543 1.5990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8543 -0.6516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8543 -0.6516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5479 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5479 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2626 1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2626 1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9773 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9773 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9773 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9773 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2626 2.8367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2626 2.8367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5479 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5479 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6589 1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6589 1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3439 -0.1057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3439 -0.1057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2567 -0.8298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2567 -0.8298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7764 2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7764 2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8156 -1.3703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8156 -1.3703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0810 -0.9462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0810 -0.9462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3140 -1.7617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3140 -1.7617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0810 -2.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0810 -2.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8156 -3.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8156 -3.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5825 -2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5825 -2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7502 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7502 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0419 -2.6106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0419 -2.6106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6229 -3.4129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6229 -3.4129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3974 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3974 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1266 -0.6256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1266 -0.6256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8215 -0.1161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8215 -0.1161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5678 0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5678 0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8781 0.4908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8781 0.4908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2954 -0.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2954 -0.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5425 -0.7634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5425 -0.7634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2305 -0.3231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2305 -0.3231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6589 -0.8023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6589 -0.8023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2626 3.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2626 3.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0683 -3.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0683 -3.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1919 -3.6347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1919 -3.6347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 16 40 1 0 0 0 0 | + | 16 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 46 0.0127 1.0794 | + | M SBV 1 46 0.0127 1.0794 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0003 | + | ID FL5FACGL0003 |
| − | FORMULA C26H28O16 | + | FORMULA C26H28O16 |
| − | EXACTMASS 596.137734848 | + | EXACTMASS 596.137734848 |
| − | AVERAGEMASS 596.49092 | + | AVERAGEMASS 596.49092 |
| − | SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O | + | SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.9579 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9579 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2567 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5555 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5555 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2567 1.5990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8543 -0.0204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1530 0.3845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1530 1.1941 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8543 1.5990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8543 -0.6516 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5479 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2626 1.1863 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9773 1.5989 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9773 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2626 2.8367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5479 2.4241 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6589 1.5989 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3439 -0.1057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2567 -0.8298 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7764 2.8855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8156 -1.3703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0810 -0.9462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3140 -1.7617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0810 -2.5823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8156 -3.0066 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5825 -2.1909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7502 -0.7283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0419 -2.6106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6229 -3.4129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3974 -0.0589 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1266 -0.6256 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8215 -0.1161 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5678 0.0925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8781 0.4908 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2954 -0.0493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5425 -0.7634 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2305 -0.3231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6589 -0.8023 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2626 3.6617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0683 -3.6617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1919 -3.6347 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 19 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
28 31 1 0 0 0 0
34 39 1 0 0 0 0
16 40 1 0 0 0 0
41 42 1 0 0 0 0
25 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ CH2OH
M SBV 1 46 0.0127 1.0794
S SKP 5
ID FL5FACGL0003
FORMULA C26H28O16
EXACTMASS 596.137734848
AVERAGEMASS 596.49092
SMILES c(c5O)c(c(c(c5)1)C(C(OC(C(OC(C4O)OCC(C4O)O)3)OC(CO)C(C3O)O)=C(c(c2)ccc(O)c2O)O1)=O)O
M END