Mol:FL5FACGS0041
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 52 57 0 0 0 0 0 0 0 0999 V2000 | + | 52 57 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1641 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1641 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1641 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1641 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4497 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4497 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7352 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7352 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7352 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7352 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4497 1.9561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4497 1.9561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0207 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0207 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6937 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6937 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6937 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6937 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0207 1.9561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0207 1.9561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0207 -0.3370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0207 -0.3370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5934 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5934 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3215 1.6945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3215 1.6945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0497 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0497 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0497 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0497 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3215 3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3215 3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5934 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5934 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4497 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4497 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8290 3.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8290 3.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4172 0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4172 0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3215 4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3215 4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0175 2.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0175 2.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1773 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1773 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9841 -1.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9841 -1.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7282 -1.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7282 -1.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9841 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9841 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1773 0.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1773 0.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4333 -0.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4333 -0.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3209 -0.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3209 -0.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2635 -2.2355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2635 -2.2355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7908 -2.6943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7908 -2.6943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3611 -1.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3611 -1.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2629 -3.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2629 -3.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8214 -3.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8214 -3.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4290 -3.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4290 -3.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0573 -3.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0573 -3.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5192 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5192 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8950 -3.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8950 -3.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2086 -3.3758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2086 -3.3758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3016 -3.6968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3016 -3.6968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1129 -3.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1129 -3.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6918 -3.4276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6918 -3.4276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2317 -4.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2317 -4.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5693 -3.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5693 -3.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9300 -3.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9300 -3.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3946 -3.3057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3946 -3.3057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0713 -3.5488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0713 -3.5488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2200 -3.6247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2200 -3.6247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8502 -4.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8502 -4.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0562 -4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0562 -4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5626 -3.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5626 -3.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8290 -2.8510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8290 -2.8510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 1 22 1 0 0 0 0 | + | 1 22 1 0 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 27 20 1 0 0 0 0 | + | 27 20 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 45 44 1 1 0 0 0 | + | 45 44 1 1 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 39 1 0 0 0 0 | + | 45 39 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 57 0.4913 -0.4835 | + | M SBV 1 57 0.4913 -0.4835 |
| − | S SKP 5 | + | S SKP 5 |
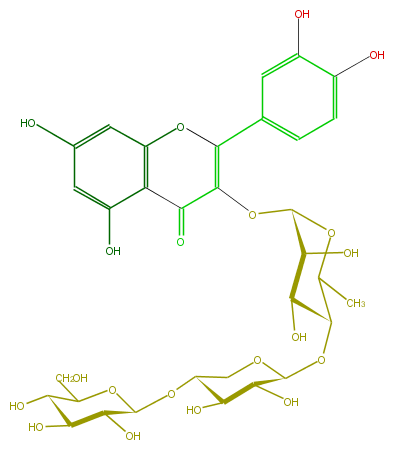
| − | ID FL5FACGS0041 | + | ID FL5FACGS0041 |
| − | FORMULA C32H38O20 | + | FORMULA C32H38O20 |
| − | EXACTMASS 742.1956436559999 | + | EXACTMASS 742.1956436559999 |
| − | AVERAGEMASS 742.63212 | + | AVERAGEMASS 742.63212 |
| − | SMILES OC(C2OC(C(O)3)C(OC(OC(=C4c(c6)ccc(O)c6O)C(c(c(O)5)c(cc(c5)O)O4)=O)C3O)C)C(C(CO2)OC(O1)C(C(O)C(O)C1CO)O)O | + | SMILES OC(C2OC(C(O)3)C(OC(OC(=C4c(c6)ccc(O)c6O)C(c(c(O)5)c(cc(c5)O)O4)=O)C3O)C)C(C(CO2)OC(O1)C(C(O)C(O)C1CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
52 57 0 0 0 0 0 0 0 0999 V2000
-2.1641 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1641 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4497 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7352 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7352 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4497 1.9561 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0207 0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6937 0.7187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6937 1.5436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0207 1.9561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0207 -0.3370 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5934 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3215 1.6945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0497 2.1150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0497 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3215 3.3762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5934 2.9557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4497 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8290 3.4057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4172 0.1928 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3215 4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0175 2.0364 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1773 -1.5006 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9841 -1.9664 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7282 -1.0706 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9841 -0.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1773 0.2965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4333 -0.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3209 -0.5594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2635 -2.2355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7908 -2.6943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3611 -1.5731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2629 -3.0570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8214 -3.5751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4290 -3.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0573 -3.0885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5192 -2.7117 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8950 -3.0685 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2086 -3.3758 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3016 -3.6968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1129 -3.5528 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6918 -3.4276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2317 -4.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5693 -3.7772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9300 -3.7703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3946 -3.3057 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0713 -3.5488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2200 -3.6247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8502 -4.0512 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0562 -4.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5626 -3.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8290 -2.8510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 8 1 0 0 0 0
4 3 1 0 0 0 0
16 21 1 0 0 0 0
1 22 1 0 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 23 1 1 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
27 20 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
31 36 1 0 0 0 0
42 43 1 1 0 0 0
43 44 1 1 0 0 0
45 44 1 1 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 39 1 0 0 0 0
51 52 1 0 0 0 0
47 51 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 ^ CH2OH
M SBV 1 57 0.4913 -0.4835
S SKP 5
ID FL5FACGS0041
FORMULA C32H38O20
EXACTMASS 742.1956436559999
AVERAGEMASS 742.63212
SMILES OC(C2OC(C(O)3)C(OC(OC(=C4c(c6)ccc(O)c6O)C(c(c(O)5)c(cc(c5)O)O4)=O)C3O)C)C(C(CO2)OC(O1)C(C(O)C(O)C1CO)O)O
M END