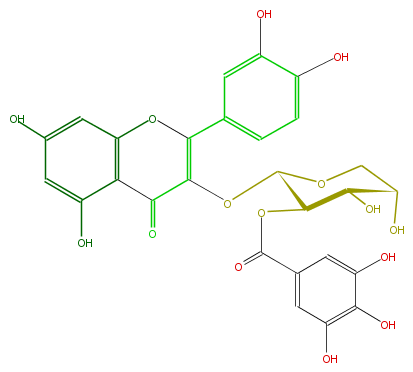
Mol:FL5FACGS0047
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1953 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1953 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1953 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1953 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4808 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4808 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7664 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7664 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7664 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7664 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4808 1.3438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4808 1.3438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0519 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0519 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3374 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3374 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3374 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3374 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0519 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0519 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0519 -0.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0519 -0.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3768 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3768 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1050 0.9232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1050 0.9232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8332 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8332 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8332 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8332 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1050 2.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1050 2.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3768 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3768 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4808 -1.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4808 -1.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6475 2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6475 2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8311 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8311 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4509 -0.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4509 -0.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1050 3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1050 3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4425 0.2674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4425 0.2674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0107 -0.4825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0107 -0.4825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8303 -0.1039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8303 -0.1039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8311 -0.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8311 -0.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1149 0.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1149 0.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3289 0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3289 0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1250 -0.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1250 -0.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2812 -0.4606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2812 -0.4606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7765 -0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7765 -0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1250 -1.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1250 -1.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6747 -1.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6747 -1.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8361 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8361 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8361 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8361 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4210 -2.7708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4210 -2.7708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0060 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0060 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0060 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0060 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4210 -1.4201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4210 -1.4201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5902 -1.4204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5902 -1.4204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5902 -2.7704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5902 -2.7704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4210 -3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4210 -3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 24 29 1 0 0 0 0 | + | 24 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 21 23 1 0 0 0 0 | + | 21 23 1 0 0 0 0 |
| − | 26 31 1 0 0 0 0 | + | 26 31 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0047 | + | ID FL5FACGS0047 |
| − | FORMULA C27H22O15 | + | FORMULA C27H22O15 |
| − | EXACTMASS 586.095870034 | + | EXACTMASS 586.095870034 |
| − | AVERAGEMASS 586.4545800000001 | + | AVERAGEMASS 586.4545800000001 |
| − | SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(OC(c(c4)cc(O)c(O)c(O)4)=O)3)OCC(C(O)3)O)Oc(c2)c(c(O)cc2O)C1=O | + | SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(OC(c(c4)cc(O)c(O)c(O)4)=O)3)OCC(C(O)3)O)Oc(c2)c(c(O)cc2O)C1=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.1953 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1953 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4808 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7664 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7664 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4808 1.3438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0519 -0.3062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3374 0.1062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3374 0.9312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0519 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0519 -0.9495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3768 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1050 0.9232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8332 1.3436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8332 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1050 2.6048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3768 2.1845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4808 -1.1310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6475 2.5877 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8311 1.3438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4509 -0.3488 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1050 3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4425 0.2674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0107 -0.4825 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8303 -0.1039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8311 -0.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1149 0.4012 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3289 0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1250 -0.5257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2812 -0.4606 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7765 -0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1250 -1.3471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6747 -1.6071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8361 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8361 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4210 -2.7708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0060 -2.4331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0060 -1.7577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4210 -1.4201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5902 -1.4204 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5902 -2.7704 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4210 -3.4454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
24 29 1 0 0 0 0
25 30 1 0 0 0 0
21 23 1 0 0 0 0
26 31 1 0 0 0 0
29 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
38 40 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
S SKP 5
ID FL5FACGS0047
FORMULA C27H22O15
EXACTMASS 586.095870034
AVERAGEMASS 586.4545800000001
SMILES c(c(O)5)cc(cc5O)C(=C1OC(C(OC(c(c4)cc(O)c(O)c(O)4)=O)3)OCC(C(O)3)O)Oc(c2)c(c(O)cc2O)C1=O
M END