Mol:FL5FACGS0049
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 35 38 0 0 0 0 0 0 0 0999 V2000 | + | 35 38 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.3093 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3093 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3093 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3093 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7530 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7530 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1967 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1967 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1967 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1967 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7530 1.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7530 1.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6404 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6404 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0841 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0841 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0841 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0841 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6404 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6404 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6404 -0.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6404 -0.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6164 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6164 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1834 1.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1834 1.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7504 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7504 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7504 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7504 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1834 2.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1834 2.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6164 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6164 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7530 -0.5622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7530 -0.5622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5440 2.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5440 2.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8042 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8042 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4792 -0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4792 -0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2638 -1.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2638 -1.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8920 -1.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8920 -1.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6927 -0.7262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6927 -0.7262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8920 -0.0244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8920 -0.0244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2638 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2638 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4631 -0.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4631 -0.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2334 -0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2334 -0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4252 -1.6632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4252 -1.6632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8920 -2.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8920 -2.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3129 -1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3129 -1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1937 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1937 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8882 -3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8882 -3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8042 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8042 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1834 3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1834 3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 30 32 1 0 0 0 0 | + | 30 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 16 35 1 0 0 0 0 | + | 16 35 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0049 | + | ID FL5FACGS0049 |
| − | KNApSAcK_ID C00005965 | + | KNApSAcK_ID C00005965 |
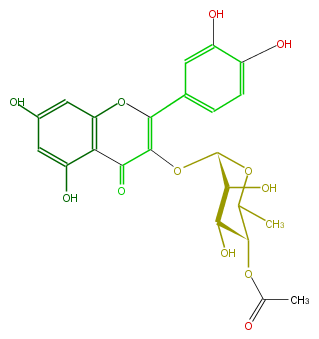
| − | NAME Quercetin 3-(4''-acetylrhamnoside) | + | NAME Quercetin 3-(4''-acetylrhamnoside) |
| − | CAS_RN 69120-16-7 | + | CAS_RN 69120-16-7 |
| − | FORMULA C23H22O12 | + | FORMULA C23H22O12 |
| − | EXACTMASS 490.111126168 | + | EXACTMASS 490.111126168 |
| − | AVERAGEMASS 490.41358 | + | AVERAGEMASS 490.41358 |
| − | SMILES c(C(=O)2)(c1O)c(OC(c(c4)cc(c(O)c4)O)=C2OC(C(O)3)OC(C(OC(C)=O)C(O)3)C)cc(c1)O | + | SMILES c(C(=O)2)(c1O)c(OC(c(c4)cc(c(O)c4)O)=C2OC(C(O)3)OC(C(OC(C)=O)C(O)3)C)cc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
35 38 0 0 0 0 0 0 0 0999 V2000
-2.3093 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3093 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7530 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1967 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1967 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7530 1.3646 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6404 0.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0841 0.4011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0841 1.0435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6404 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6404 -0.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6164 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1834 1.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7504 1.4883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7504 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1834 2.4703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6164 2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7530 -0.5622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5440 2.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8042 1.3646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4792 -0.0084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2638 -1.0610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8920 -1.4237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6927 -0.7262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8920 -0.0244 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2638 0.3383 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4631 -0.3592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2334 -0.3592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4252 -1.6632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8920 -2.0730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3129 -1.0843 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1937 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8882 -3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8042 -2.5955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1834 3.1248 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 21 1 0 0 0 0
30 32 1 0 0 0 0
32 33 2 0 0 0 0
32 34 1 0 0 0 0
16 35 1 0 0 0 0
S SKP 8
ID FL5FACGS0049
KNApSAcK_ID C00005965
NAME Quercetin 3-(4''-acetylrhamnoside)
CAS_RN 69120-16-7
FORMULA C23H22O12
EXACTMASS 490.111126168
AVERAGEMASS 490.41358
SMILES c(C(=O)2)(c1O)c(OC(c(c4)cc(c(O)c4)O)=C2OC(C(O)3)OC(C(OC(C)=O)C(O)3)C)cc(c1)O
M END