Mol:FL5FACGS0056
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 46 50 0 0 0 0 0 0 0 0999 V2000 | + | 46 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.3364 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3364 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3364 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3364 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7801 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7801 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2238 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2238 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2238 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2238 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7801 0.6155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7801 0.6155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6675 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6675 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1112 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1112 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1112 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1112 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6675 0.6155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6675 0.6155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6675 -1.1701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6675 -1.1701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4107 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4107 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8438 0.4117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8438 0.4117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2768 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2768 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2768 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2768 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8438 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8438 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4107 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4107 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7801 -1.3114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7801 -1.3114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3300 1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3300 1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8520 0.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8520 0.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4818 -0.7115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4818 -0.7115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8438 2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8438 2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4242 -0.0049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4242 -0.0049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9311 -0.4182 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9311 -0.4182 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2794 -0.4736 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.2794 -0.4736 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.2481 -0.8664 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.2481 -0.8664 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.7550 -0.4532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.7550 -0.4532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.1034 -0.3977 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.1034 -0.3977 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 0.0035 0.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0035 0.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7554 -0.9180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7554 -0.9180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5864 0.1616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5864 0.1616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5823 -1.1041 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.5823 -1.1041 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.7694 -1.7425 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7694 -1.7425 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.3900 -1.7378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.3900 -1.7378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.9139 -1.9305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.9139 -1.9305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.6789 -1.4071 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.6789 -1.4071 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 3.1110 -1.4770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1110 -1.4770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4958 -1.2799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4958 -1.2799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1732 -1.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1732 -1.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5021 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5021 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6789 -1.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6789 -1.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7935 -2.4062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7935 -2.4062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9853 -1.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9853 -1.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5027 -0.7057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5027 -0.7057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4174 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4174 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.1295 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.1295 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 28 31 1 0 0 0 0 | + | 28 31 1 0 0 0 0 |
| − | 24 21 1 0 0 0 0 | + | 24 21 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 38 27 1 0 0 0 0 | + | 38 27 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0056 | + | ID FL5FACGS0056 |
| − | KNApSAcK_ID C00005980 | + | KNApSAcK_ID C00005980 |
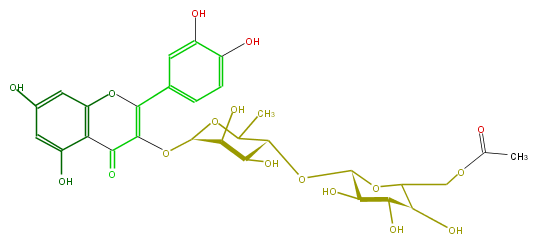
| − | NAME Quercetin 3-(6'''-acetylglucosyl)(1->4)-rhamnoside | + | NAME Quercetin 3-(6'''-acetylglucosyl)(1->4)-rhamnoside |
| − | CAS_RN 145626-31-9 | + | CAS_RN 145626-31-9 |
| − | FORMULA C29H32O17 | + | FORMULA C29H32O17 |
| − | EXACTMASS 652.163949598 | + | EXACTMASS 652.163949598 |
| − | AVERAGEMASS 652.55418 | + | AVERAGEMASS 652.55418 |
| − | SMILES [C@H]([C@@H](O)1)(O)[C@@H](C(O[C@@H]1O[C@H](C5C)[C@@H](O)[C@H](O)[C@H](O5)OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)cc(c(c2)O)O)COC(C)=O)O | + | SMILES [C@H]([C@@H](O)1)(O)[C@@H](C(O[C@@H]1O[C@H](C5C)[C@@H](O)[C@H](O)[C@H](O5)OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)cc(c(c2)O)O)COC(C)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
46 50 0 0 0 0 0 0 0 0999 V2000
-4.3364 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3364 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7801 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2238 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2238 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7801 0.6155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6675 -0.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1112 -0.3481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1112 0.2943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6675 0.6155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6675 -1.1701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4107 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8438 0.4117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2768 0.7391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2768 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8438 1.7211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4107 1.3938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7801 -1.3114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3300 1.7441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8520 0.7480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4818 -0.7115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8438 2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4242 -0.0049 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9311 -0.4182 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2794 -0.4736 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.2481 -0.8664 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.7550 -0.4532 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.1034 -0.3977 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
0.0035 0.2708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7554 -0.9180 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5864 0.1616 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5823 -1.1041 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.7694 -1.7425 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.3900 -1.7378 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.9139 -1.9305 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.6789 -1.4071 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
3.1110 -1.4770 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4958 -1.2799 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1732 -1.5485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5021 -2.3756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6789 -1.4071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7935 -2.4062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9853 -1.1276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5027 -0.7057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4174 -0.1782 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.1295 -0.7695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
25 29 1 0 0 0 0
26 30 1 0 0 0 0
28 31 1 0 0 0 0
24 21 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
36 41 1 0 0 0 0
35 42 1 0 0 0 0
38 27 1 0 0 0 0
41 43 1 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
S SKP 8
ID FL5FACGS0056
KNApSAcK_ID C00005980
NAME Quercetin 3-(6'''-acetylglucosyl)(1->4)-rhamnoside
CAS_RN 145626-31-9
FORMULA C29H32O17
EXACTMASS 652.163949598
AVERAGEMASS 652.55418
SMILES [C@H]([C@@H](O)1)(O)[C@@H](C(O[C@@H]1O[C@H](C5C)[C@@H](O)[C@H](O)[C@H](O5)OC(C(=O)3)=C(Oc(c4)c3c(cc4O)O)c(c2)cc(c(c2)O)O)COC(C)=O)O
M END