Mol:FL5FACGS0117
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | 5.5676 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5676 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8531 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8531 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8531 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8531 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5676 2.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5676 2.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2820 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2820 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.2820 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.2820 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1386 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1386 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4242 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4242 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7097 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7097 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7097 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7097 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4242 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4242 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1386 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1386 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9952 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9952 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2807 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2807 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2807 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2807 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9952 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9952 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4242 -0.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4242 -0.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5663 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5663 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7376 0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7376 0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9952 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9952 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.8739 2.9154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.8739 2.9154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5676 3.7119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5676 3.7119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5009 -1.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5009 -1.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1614 -2.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1614 -2.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8131 -1.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8131 -1.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6318 -1.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6318 -1.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9714 -1.0593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9714 -1.0593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3196 -1.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3196 -1.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9448 -1.0887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9448 -1.0887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3947 -1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3947 -1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2467 -2.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2467 -2.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6470 -2.1024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6470 -2.1024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9072 -2.3135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9072 -2.3135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3251 -3.4110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3251 -3.4110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9856 -3.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9856 -3.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6374 -3.3995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6374 -3.3995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4561 -3.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4561 -3.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7956 -2.8011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7956 -2.8011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1438 -3.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1438 -3.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7691 -2.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7691 -2.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2190 -2.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2190 -2.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0710 -4.0453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0710 -4.0453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4712 -3.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4712 -3.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7314 -4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7314 -4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0524 1.9274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0524 1.9274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6397 1.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6397 1.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8414 1.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8414 1.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0479 1.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0479 1.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4605 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4605 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2589 1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2589 1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4159 2.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4159 2.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8957 2.5635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8957 2.5635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5356 1.4442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5356 1.4442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0884 1.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0884 1.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0129 0.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0129 0.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3403 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3403 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9284 4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9284 4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1642 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1642 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5761 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5761 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4000 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4000 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8125 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8125 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6375 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6375 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.0500 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.0500 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6375 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6375 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8125 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8125 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.8739 2.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.8739 2.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.2161 3.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.2161 3.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 33 1 0 0 0 0 | + | 37 33 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 55 1 0 0 0 0 | + | 47 55 1 0 0 0 0 |
| − | 48 18 1 0 0 0 0 | + | 48 18 1 0 0 0 0 |
| − | 56 57 2 0 0 0 0 | + | 56 57 2 0 0 0 0 |
| − | 56 58 1 0 0 0 0 | + | 56 58 1 0 0 0 0 |
| − | 58 59 2 0 0 0 0 | + | 58 59 2 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 2 0 0 0 0 | + | 60 61 2 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 2 0 0 0 0 | + | 62 63 2 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 64 65 2 0 0 0 0 | + | 64 65 2 0 0 0 0 |
| − | 65 60 1 0 0 0 0 | + | 65 60 1 0 0 0 0 |
| − | 63 66 1 0 0 0 0 | + | 63 66 1 0 0 0 0 |
| − | 52 56 1 0 0 0 0 | + | 52 56 1 0 0 0 0 |
| − | 62 67 1 0 0 0 0 | + | 62 67 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0117 | + | ID FL5FACGS0117 |
| − | KNApSAcK_ID C00013889 | + | KNApSAcK_ID C00013889 |
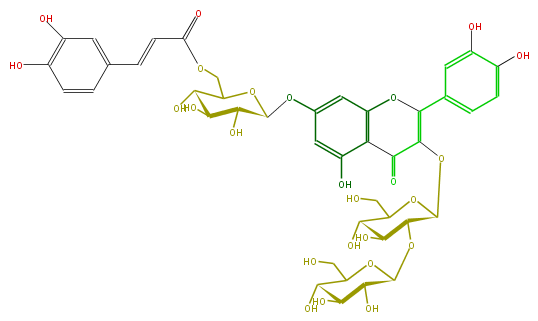
| − | NAME Quercetin 3-sophoroside-7-(6''-(E)-caffeoylglucoside);2-(3,4-Dihydroxyphenyl)-7-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3-sophoroside-7-(6''-(E)-caffeoylglucoside);2-(3,4-Dihydroxyphenyl)-7-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 247576-99-4 | + | CAS_RN 247576-99-4 |
| − | FORMULA C42H46O25 | + | FORMULA C42H46O25 |
| − | EXACTMASS 950.2328170220001 | + | EXACTMASS 950.2328170220001 |
| − | AVERAGEMASS 950.79964 | + | AVERAGEMASS 950.79964 |
| − | SMILES Oc(c1)c(O)ccc1C(O4)=C(C(=O)c(c(O)5)c(cc(OC(O6)C(C(O)C(O)C(COC(=O)C=Cc(c7)cc(O)c(O)c7)6)O)c5)4)OC(O3)C(C(C(C(CO)3)O)O)OC(C2O)OC(C(C2O)O)CO | + | SMILES Oc(c1)c(O)ccc1C(O4)=C(C(=O)c(c(O)5)c(cc(OC(O6)C(C(O)C(O)C(COC(=O)C=Cc(c7)cc(O)c(O)c7)6)O)c5)4)OC(O3)C(C(C(C(CO)3)O)O)OC(C2O)OC(C(C2O)O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
5.5676 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8531 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8531 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5676 2.9861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2820 2.5736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.2820 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1386 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4242 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7097 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7097 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4242 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1386 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9952 1.7486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2807 1.3361 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2807 0.5111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9952 0.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4242 -0.5621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5663 1.7486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7376 0.0656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9952 -0.6280 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.8739 2.9154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5676 3.7119 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5009 -1.6693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1614 -2.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8131 -1.6577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6318 -1.5542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9714 -1.0593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3196 -1.5656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9448 -1.0887 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3947 -1.0228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2467 -2.3036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6470 -2.1024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9072 -2.3135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3251 -3.4110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9856 -3.9058 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6374 -3.3995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4561 -3.2959 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7956 -2.8011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1438 -3.3073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7691 -2.8305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2190 -2.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0710 -4.0453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4712 -3.8441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7314 -4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0524 1.9274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6397 1.2127 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8414 1.4218 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0479 1.1950 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4605 1.9097 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2589 1.7007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4159 2.2865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8957 2.5635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5356 1.4442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0884 1.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0129 0.7819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3403 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9284 4.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1642 3.3418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5761 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4000 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8125 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6375 3.3427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.0500 2.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6375 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8125 1.9138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.8739 2.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.2161 3.9213 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 33 1 0 0 0 0
26 19 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
51 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 55 1 0 0 0 0
48 18 1 0 0 0 0
56 57 2 0 0 0 0
56 58 1 0 0 0 0
58 59 2 0 0 0 0
59 60 1 0 0 0 0
60 61 2 0 0 0 0
61 62 1 0 0 0 0
62 63 2 0 0 0 0
63 64 1 0 0 0 0
64 65 2 0 0 0 0
65 60 1 0 0 0 0
63 66 1 0 0 0 0
52 56 1 0 0 0 0
62 67 1 0 0 0 0
S SKP 8
ID FL5FACGS0117
KNApSAcK_ID C00013889
NAME Quercetin 3-sophoroside-7-(6''-(E)-caffeoylglucoside);2-(3,4-Dihydroxyphenyl)-7-[[6-O-[(2E)-3-(3,4-dihydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-3-[(2-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-4H-1-benzopyran-4-one
CAS_RN 247576-99-4
FORMULA C42H46O25
EXACTMASS 950.2328170220001
AVERAGEMASS 950.79964
SMILES Oc(c1)c(O)ccc1C(O4)=C(C(=O)c(c(O)5)c(cc(OC(O6)C(C(O)C(O)C(COC(=O)C=Cc(c7)cc(O)c(O)c7)6)O)c5)4)OC(O3)C(C(C(C(CO)3)O)O)OC(C2O)OC(C(C2O)O)CO
M END