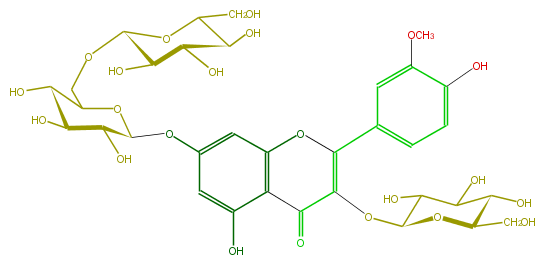
Mol:FL5FADGL0031
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 56 61 0 0 0 0 0 0 0 0999 V2000 | + | 56 61 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5096 -0.8342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5096 -0.8342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5096 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5096 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7950 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7950 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0805 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0805 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0805 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0805 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7951 -0.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7951 -0.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6340 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6340 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3486 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3486 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3485 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3485 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6340 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6340 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6340 -2.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6340 -2.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2483 -0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2483 -0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9765 -0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9765 -0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7047 -0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7047 -0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7047 0.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7047 0.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9765 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9765 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2483 0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2483 0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7951 -2.8964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7951 -2.8964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3442 1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3442 1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1454 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1454 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0721 -2.1851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0721 -2.1851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6645 0.5703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6645 0.5703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3041 -0.3118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3041 -0.3118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5663 -0.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5663 -0.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9401 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9401 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2283 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2283 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9748 0.1054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9748 0.1054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2848 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2848 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8205 -0.1346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8205 -0.1346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1673 -0.9459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1673 -0.9459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2109 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2109 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1093 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1093 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4779 1.0346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4779 1.0346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8732 1.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8732 1.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8671 1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8671 1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5263 2.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5263 2.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2066 1.5832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2066 1.5832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8535 1.2114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8535 1.2114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1930 0.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1930 0.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2314 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2314 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4524 1.7210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4524 1.7210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1719 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1719 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7856 -2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7856 -2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5286 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5286 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2762 -2.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2762 -2.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6625 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6625 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9195 -1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9195 -1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6285 -1.7855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6285 -1.7855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3042 -1.3946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3042 -1.3946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2848 -1.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2848 -1.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9779 1.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9779 1.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5449 2.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5449 2.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9916 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9916 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9916 -3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9916 -3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1550 3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1550 3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 42 1 0 0 0 0 | + | 47 42 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 43 21 1 0 0 0 0 | + | 43 21 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 16 51 1 0 0 0 0 | + | 16 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 36 55 1 0 0 0 0 | + | 36 55 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 57 -0.0014 -0.6725 | + | M SBV 1 57 -0.0014 -0.6725 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 59 -0.7154 -0.1089 | + | M SBV 2 59 -0.7154 -0.1089 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 55 56 | + | M SAL 3 2 55 56 |
| − | M SBL 3 1 61 | + | M SBL 3 1 61 |
| − | M SMT 3 CH2OH | + | M SMT 3 CH2OH |
| − | M SBV 3 61 -0.7070 -0.0868 | + | M SBV 3 61 -0.7070 -0.0868 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGL0031 | + | ID FL5FADGL0031 |
| − | FORMULA C34H42O22 | + | FORMULA C34H42O22 |
| − | EXACTMASS 802.216773028 | + | EXACTMASS 802.216773028 |
| − | AVERAGEMASS 802.68408 | + | AVERAGEMASS 802.68408 |
| − | SMILES OC(C6O)C(OC(CO)C6O)OC(C2=O)=C(Oc(c3)c2c(cc3OC(O4)C(C(O)C(O)C4COC(O5)C(O)C(O)C(C5CO)O)O)O)c(c1)cc(c(c1)O)OC | + | SMILES OC(C6O)C(OC(CO)C6O)OC(C2=O)=C(Oc(c3)c2c(cc3OC(O4)C(C(O)C(O)C4COC(O5)C(O)C(O)C(C5CO)O)O)O)c(c1)cc(c(c1)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
56 61 0 0 0 0 0 0 0 0999 V2000
-1.5096 -0.8342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5096 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7950 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0805 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0805 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7951 -0.4216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6340 -2.0716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3486 -1.6591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3485 -0.8341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6340 -0.4216 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6340 -2.7149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2483 -0.2626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9765 -0.6832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7047 -0.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7047 0.5781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9765 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2483 0.5782 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7951 -2.8964 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3442 1.0126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1454 -0.4215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0721 -2.1851 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6645 0.5703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3041 -0.3118 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5663 -0.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9401 -0.5160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2283 0.1021 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9748 0.1054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2848 0.4595 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8205 -0.1346 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1673 -0.9459 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2109 0.5102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1093 1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4779 1.0346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8732 1.4165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8671 1.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5263 2.0219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2066 1.5832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8535 1.2114 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1930 0.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2314 0.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4524 1.7210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1719 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7856 -2.3979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5286 -2.1855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2762 -2.3978 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6625 -1.7285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9195 -1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6285 -1.7855 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3042 -1.3946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2848 -1.8882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9779 1.6712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5449 2.8237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9916 -2.2890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9916 -3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8193 2.1087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1550 3.3486 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
38 31 1 0 0 0 0
35 41 1 0 0 0 0
25 20 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 42 1 0 0 0 0
42 48 1 0 0 0 0
47 49 1 0 0 0 0
46 50 1 0 0 0 0
43 21 1 0 0 0 0
51 52 1 0 0 0 0
16 51 1 0 0 0 0
53 54 1 0 0 0 0
45 53 1 0 0 0 0
55 56 1 0 0 0 0
36 55 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 OCH3
M SBV 1 57 -0.0014 -0.6725
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 59
M SMT 2 CH2OH
M SBV 2 59 -0.7154 -0.1089
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 55 56
M SBL 3 1 61
M SMT 3 CH2OH
M SBV 3 61 -0.7070 -0.0868
S SKP 5
ID FL5FADGL0031
FORMULA C34H42O22
EXACTMASS 802.216773028
AVERAGEMASS 802.68408
SMILES OC(C6O)C(OC(CO)C6O)OC(C2=O)=C(Oc(c3)c2c(cc3OC(O4)C(C(O)C(O)C4COC(O5)C(O)C(O)C(C5CO)O)O)O)c(c1)cc(c(c1)O)OC
M END