Mol:FL5FAGGS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 33 36 0 0 0 0 0 0 0 0999 V2000 | + | 33 36 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.0021 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0021 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0021 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0021 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4458 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4458 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8895 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8895 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8895 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8895 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4458 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4458 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3332 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3332 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2231 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2231 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2231 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2231 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3332 0.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3332 0.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3332 -0.8752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3332 -0.8752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7792 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7792 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3462 0.5829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3462 0.5829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9131 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9131 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9131 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9131 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3462 1.8923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3462 1.8923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7792 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7792 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5582 0.9102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5582 0.9102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4458 -1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4458 -1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7792 -0.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7792 -0.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4973 1.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4973 1.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4800 0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4800 0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5091 -1.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5091 -1.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1373 -1.9811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1373 -1.9811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9380 -1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9380 -1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1373 -0.5819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1373 -0.5819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5091 -0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5091 -0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7084 -0.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7084 -0.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4787 -0.9166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4787 -0.9166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6705 -2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6705 -2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9857 -2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9857 -2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5582 -1.6417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5582 -1.6417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3462 2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3462 2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 14 22 1 0 0 0 0 | + | 14 22 1 0 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 27 20 1 0 0 0 0 | + | 27 20 1 0 0 0 0 |
| − | 16 33 1 0 0 0 0 | + | 16 33 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAGGS0004 | + | ID FL5FAGGS0004 |
| − | KNApSAcK_ID C00005730 | + | KNApSAcK_ID C00005730 |
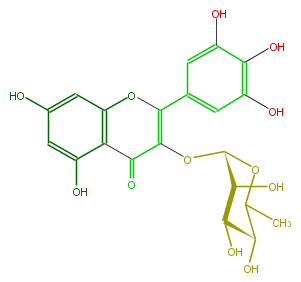
| − | NAME Myricitrin | + | NAME Myricitrin |
| − | CAS_RN 17912-87-7 | + | CAS_RN 17912-87-7 |
| − | FORMULA C21H20O12 | + | FORMULA C21H20O12 |
| − | EXACTMASS 464.095476104 | + | EXACTMASS 464.095476104 |
| − | AVERAGEMASS 464.37629999999996 | + | AVERAGEMASS 464.37629999999996 |
| − | SMILES C(=C3OC(O4)C(O)C(O)C(O)C(C)4)(Oc(c2)c(C3=O)c(cc(O)2)O)c(c1)cc(O)c(O)c(O)1 | + | SMILES C(=C3OC(O4)C(O)C(O)C(O)C(C)4)(Oc(c2)c(C3=O)c(cc(O)2)O)c(c1)cc(O)c(O)c(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
33 36 0 0 0 0 0 0 0 0999 V2000
-2.0021 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0021 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4458 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8895 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8895 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4458 0.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3332 -0.3744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2231 -0.0532 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2231 0.5892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3332 0.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3332 -0.8752 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7792 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3462 0.5829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9131 0.9102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9131 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3462 1.8923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7792 1.5649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5582 0.9102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4458 -1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7792 -0.3743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4973 1.8838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4800 0.5830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5091 -1.6184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1373 -1.9811 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9380 -1.2836 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1373 -0.5819 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5091 -0.2191 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7084 -0.9166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4787 -0.9166 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6705 -2.2206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9857 -2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5582 -1.6417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3462 2.5468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
15 21 1 0 0 0 0
14 22 1 0 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 23 1 1 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
27 20 1 0 0 0 0
16 33 1 0 0 0 0
S SKP 8
ID FL5FAGGS0004
KNApSAcK_ID C00005730
NAME Myricitrin
CAS_RN 17912-87-7
FORMULA C21H20O12
EXACTMASS 464.095476104
AVERAGEMASS 464.37629999999996
SMILES C(=C3OC(O4)C(O)C(O)C(O)C(C)4)(Oc(c2)c(C3=O)c(cc(O)2)O)c(c1)cc(O)c(O)c(O)1
M END