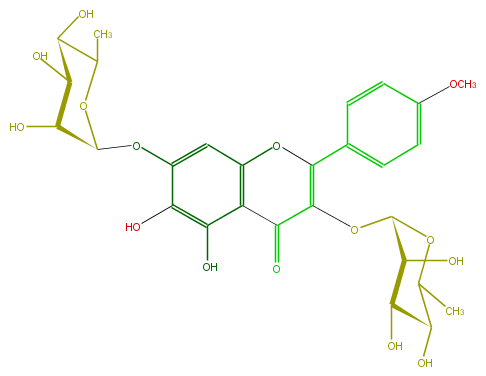
Mol:FL5FEAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7577 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7577 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7577 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7577 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0566 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0566 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3556 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3556 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3556 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3556 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0566 0.8527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0566 0.8527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3455 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3455 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0465 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0465 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0465 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0465 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3455 0.8527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3455 0.8527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3455 -1.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3455 -1.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7472 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7472 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4617 0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4617 0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1762 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1762 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1762 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1762 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4617 2.0901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4617 2.0901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7472 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7472 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4584 0.8526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4584 0.8526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8980 -0.8229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8980 -0.8229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0566 -1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0566 -1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4584 -0.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4584 -0.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6373 -2.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6373 -2.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4290 -2.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4290 -2.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1778 -1.9193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1778 -1.9193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4290 -1.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4290 -1.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6373 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6373 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8885 -1.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8885 -1.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8592 -1.4569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8592 -1.4569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6359 -3.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6359 -3.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2394 -3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2394 -3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7957 -2.4545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7957 -2.4545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0500 1.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0500 1.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2584 0.7665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2584 0.7665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5097 1.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5097 1.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2584 2.5297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2584 2.5297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0500 2.9868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0500 2.9868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7990 2.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7990 2.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5451 3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5451 3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2584 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2584 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4684 2.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4684 2.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7794 1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7794 1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8790 2.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8790 2.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7794 1.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7794 1.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 2 21 1 0 0 0 0 | + | 2 21 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 22 29 1 0 0 0 0 | + | 22 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 33 18 1 0 0 0 0 | + | 33 18 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 42 43 | + | M SAL 1 2 42 43 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 47 -0.7029 -0.4058 | + | M SBV 1 47 -0.7029 -0.4058 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FEAGS0011 | + | ID FL5FEAGS0011 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES Oc(c4O)c(c2cc(OC(O5)C(O)C(O)C(O)C(C)5)4)C(C(=C(c(c3)ccc(OC)c3)O2)OC(O1)C(C(O)C(C1C)O)O)=O | + | SMILES Oc(c4O)c(c2cc(OC(O5)C(O)C(O)C(O)C(C)5)4)C(C(=C(c(c3)ccc(OC)c3)O2)OC(O1)C(C(O)C(C1C)O)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.7577 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7577 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0566 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3556 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3556 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0566 0.8527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3455 -0.7662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0465 -0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0465 0.4480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3455 0.8527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3455 -1.6113 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7472 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4617 0.4400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1762 0.8526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1762 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4617 2.0901 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7472 1.6776 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4584 0.8526 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8980 -0.8229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0566 -1.5753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4584 -0.7660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6373 -2.3412 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4290 -2.7983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1778 -1.9193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4290 -1.0351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6373 -0.5779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8885 -1.4569 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8592 -1.4569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6359 -3.1427 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2394 -3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7957 -2.4545 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0500 1.2235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2584 0.7665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5097 1.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2584 2.5297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0500 2.9868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7990 2.1079 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5451 3.4919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2584 3.1001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4684 2.6479 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7794 1.2286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8790 2.0834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7794 1.5636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
2 21 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
27 28 1 0 0 0 0
22 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
26 19 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
33 18 1 0 0 0 0
42 43 1 0 0 0 0
15 42 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 42 43
M SBL 1 1 47
M SMT 1 OCH3
M SBV 1 47 -0.7029 -0.4058
S SKP 5
ID FL5FEAGS0011
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES Oc(c4O)c(c2cc(OC(O5)C(O)C(O)C(O)C(C)5)4)C(C(=C(c(c3)ccc(OC)c3)O2)OC(O1)C(C(O)C(C1C)O)O)=O
M END