Mol:FL5FECGS0027
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 38 41 0 0 0 0 0 0 0 0999 V2000 | + | 38 41 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.5284 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5284 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5284 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5284 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8139 -2.0229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8139 -2.0229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0995 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0995 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0994 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0994 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8140 -0.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8140 -0.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3849 -2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3849 -2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6705 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6705 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6704 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6704 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3850 -0.3729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3850 -0.3729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3849 -2.7742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3849 -2.7742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0438 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0438 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7720 -0.7935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7720 -0.7935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5002 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5002 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5001 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5001 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7718 0.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7718 0.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0437 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0437 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2426 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2426 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8139 -2.7328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8139 -2.7328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2204 0.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2204 0.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2520 0.2071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2520 0.2071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4911 0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4911 0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1312 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1312 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4491 -1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4491 -1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2101 -0.9347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2101 -0.9347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5700 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5700 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7692 0.6942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7692 0.6942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2375 -0.5444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2375 -0.5444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3640 -1.5629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3640 -1.5629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1132 -1.9481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1132 -1.9481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0308 -1.4183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0308 -1.4183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7718 1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7718 1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3389 2.7742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3389 2.7742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0640 -2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0640 -2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1763 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1763 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0704 -1.9423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0704 -1.9423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0308 -2.3901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0308 -2.3901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2024 -2.5501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2024 -2.5501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 20 1 0 0 0 0 | + | 22 20 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 2 30 1 0 0 0 0 | + | 2 30 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 8 34 1 0 0 0 0 | + | 8 34 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 24 36 1 0 0 0 0 | + | 24 36 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 30 31 | + | M SAL 1 2 30 31 |
| − | M SBL 1 1 34 | + | M SBL 1 1 34 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 34 0.5848 0.3376 | + | M SBV 1 34 0.5848 0.3376 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 32 33 | + | M SAL 2 2 32 33 |
| − | M SBL 2 1 36 | + | M SBL 2 1 36 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 36 0.0000 -0.7336 | + | M SBV 2 36 0.0000 -0.7336 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 34 35 | + | M SAL 3 2 34 35 |
| − | M SBL 3 1 38 | + | M SBL 3 1 38 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 38 -0.7345 0.4241 | + | M SBV 3 38 -0.7345 0.4241 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 3 36 37 38 | + | M SAL 4 3 36 37 38 |
| − | M SBL 4 1 41 | + | M SBL 4 1 41 |
| − | M SMT 4 COOH | + | M SMT 4 COOH |
| − | M SBV 4 41 -0.6213 0.8735 | + | M SBV 4 41 -0.6213 0.8735 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FECGS0027 | + | ID FL5FECGS0027 |
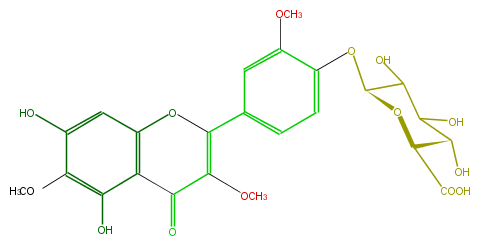
| − | FORMULA C24H24O14 | + | FORMULA C24H24O14 |
| − | EXACTMASS 536.116605476 | + | EXACTMASS 536.116605476 |
| − | AVERAGEMASS 536.43896 | + | AVERAGEMASS 536.43896 |
| − | SMILES c(c(O)4)(c(O)cc(c43)OC(=C(OC)C(=O)3)c(c2)ccc(c(OC)2)OC(C(O)1)OC(C(O)=O)C(C1O)O)OC | + | SMILES c(c(O)4)(c(O)cc(c43)OC(=C(OC)C(=O)3)c(c2)ccc(c(OC)2)OC(C(O)1)OC(C(O)=O)C(C1O)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
38 41 0 0 0 0 0 0 0 0999 V2000
-3.5284 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5284 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8139 -2.0229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0995 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0994 -0.7854 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8140 -0.3728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3849 -2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6705 -1.6104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6704 -0.7855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3850 -0.3729 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3849 -2.7742 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0438 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7720 -0.7935 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5002 -0.3730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5001 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7718 0.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0437 0.4678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2426 -0.3730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8139 -2.7328 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2204 0.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2520 0.2071 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4911 0.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1312 -0.3597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4491 -1.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2101 -0.9347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5700 -0.5019 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7692 0.6942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2375 -0.5444 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3640 -1.5629 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1132 -1.9481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0308 -1.4183 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7718 1.6217 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3389 2.7742 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0640 -2.0345 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1763 -2.6765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0704 -1.9423 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0308 -2.3901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2024 -2.5501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 20 1 0 0 0 0
30 31 1 0 0 0 0
2 30 1 0 0 0 0
32 33 1 0 0 0 0
16 32 1 0 0 0 0
34 35 1 0 0 0 0
8 34 1 0 0 0 0
36 37 2 0 0 0 0
36 38 1 0 0 0 0
24 36 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 30 31
M SBL 1 1 34
M SMT 1 ^ OCH3
M SBV 1 34 0.5848 0.3376
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 32 33
M SBL 2 1 36
M SMT 2 OCH3
M SBV 2 36 0.0000 -0.7336
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 34 35
M SBL 3 1 38
M SMT 3 OCH3
M SBV 3 38 -0.7345 0.4241
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 3 36 37 38
M SBL 4 1 41
M SMT 4 COOH
M SBV 4 41 -0.6213 0.8735
S SKP 5
ID FL5FECGS0027
FORMULA C24H24O14
EXACTMASS 536.116605476
AVERAGEMASS 536.43896
SMILES c(c(O)4)(c(O)cc(c43)OC(=C(OC)C(=O)3)c(c2)ccc(c(OC)2)OC(C(O)1)OC(C(O)=O)C(C1O)O)OC
M END