Mol:FL5FECNSS008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 30 32 0 0 0 0 0 0 0 0999 V2000 | + | 30 32 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.0268 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0268 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5830 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5830 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5831 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5831 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0267 -1.9888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0267 -1.9888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4705 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4705 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4705 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4705 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0268 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0268 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4704 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4704 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0858 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0858 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0858 -1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0858 -1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4605 -2.8816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4605 -2.8816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6419 -2.9522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6419 -2.9522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6419 -3.6070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6419 -3.6070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2089 -3.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2089 -3.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7759 -3.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7759 -3.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7759 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7759 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2089 -2.6249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2089 -2.6249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0522 -1.9392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0522 -1.9392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6419 -4.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6419 -4.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4459 -3.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4459 -3.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2141 -4.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2141 -4.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1005 -4.1941 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1005 -4.1941 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3234 -3.9494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3234 -3.9494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5713 -4.4660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5713 -4.4660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7415 -0.3226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7415 -0.3226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8846 -1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8846 -1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3660 -2.8626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3660 -2.8626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3547 -2.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3547 -2.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7077 -0.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7077 -0.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2494 0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2494 0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 18 3 1 0 0 0 0 | + | 18 3 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 22 23 2 0 0 0 0 | + | 22 23 2 0 0 0 0 |
| − | 22 24 2 0 0 0 0 | + | 22 24 2 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 2 25 1 0 0 0 0 | + | 2 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 16 27 1 0 0 0 0 | + | 16 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 1 29 1 0 0 0 0 | + | 1 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 29 30 | + | M SAL 3 2 29 30 |
| − | M SBL 3 1 31 | + | M SBL 3 1 31 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 31 -2.2053 0.409 | + | M SVB 3 31 -2.2053 0.409 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 27 28 | + | M SAL 2 2 27 28 |
| − | M SBL 2 1 29 | + | M SBL 2 1 29 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 29 1.7367 1.6823 | + | M SVB 2 29 1.7367 1.6823 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 25 26 | + | M SAL 1 2 25 26 |
| − | M SBL 1 1 27 | + | M SBL 1 1 27 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 27 -2.5435 -0.7276 | + | M SVB 1 27 -2.5435 -0.7276 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FECNSS008 | + | ID FL5FECNSS008 |
| − | KNApSAcK_ID C00004984 | + | KNApSAcK_ID C00004984 |
| − | NAME Veronicafolin 3-O-sulfate | + | NAME Veronicafolin 3-O-sulfate |
| − | CAS_RN 74545-45-2 | + | CAS_RN 74545-45-2 |
| − | FORMULA C18H16O11S | + | FORMULA C18H16O11S |
| − | EXACTMASS 440.041332044 | + | EXACTMASS 440.041332044 |
| − | AVERAGEMASS 440.37904000000003 | + | AVERAGEMASS 440.37904000000003 |
| − | SMILES c(c3OC)c(O1)c(c(c3OC)O)C(C(OS(O)(=O)=O)=C1c(c2)cc(OC)c(O)c2)=O | + | SMILES c(c3OC)c(O1)c(c(c3OC)O)C(C(OS(O)(=O)=O)=C1c(c2)cc(OC)c(O)c2)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
30 32 0 0 0 0 0 0 0 0999 V2000
-1.0268 -0.7041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5830 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5831 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0267 -1.9888 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4705 -1.6676 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4705 -1.0253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0268 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4704 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0858 -2.6312 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0858 -1.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4605 -2.8816 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6419 -2.9522 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6419 -3.6070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2089 -3.9342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7759 -3.6069 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7759 -2.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2089 -2.6249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0522 -1.9392 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6419 -4.1068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4459 -3.6077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2141 -4.7508 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1005 -4.1941 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
0.3234 -3.9494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5713 -4.4660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7415 -0.3226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8846 -1.1351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3660 -2.8626 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3547 -2.7124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7077 -0.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2494 0.8034 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
18 3 1 0 0 0 0
19 15 1 0 0 0 0
22 23 2 0 0 0 0
22 24 2 0 0 0 0
21 22 1 0 0 0 0
20 22 1 0 0 0 0
8 20 1 0 0 0 0
2 25 1 0 0 0 0
25 26 1 0 0 0 0
16 27 1 0 0 0 0
27 28 1 0 0 0 0
1 29 1 0 0 0 0
29 30 1 0 0 0 0
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 29 30
M SBL 3 1 31
M SMT 3 OCH3
M SVB 3 31 -2.2053 0.409
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 27 28
M SBL 2 1 29
M SMT 2 OCH3
M SVB 2 29 1.7367 1.6823
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 25 26
M SBL 1 1 27
M SMT 1 OCH3
M SVB 1 27 -2.5435 -0.7276
S SKP 8
ID FL5FECNSS008
KNApSAcK_ID C00004984
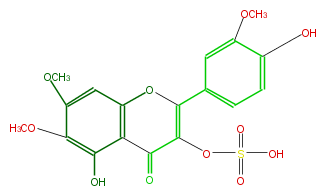
NAME Veronicafolin 3-O-sulfate
CAS_RN 74545-45-2
FORMULA C18H16O11S
EXACTMASS 440.041332044
AVERAGEMASS 440.37904000000003
SMILES c(c3OC)c(O1)c(c(c3OC)O)C(C(OS(O)(=O)=O)=C1c(c2)cc(OC)c(O)c2)=O
M END