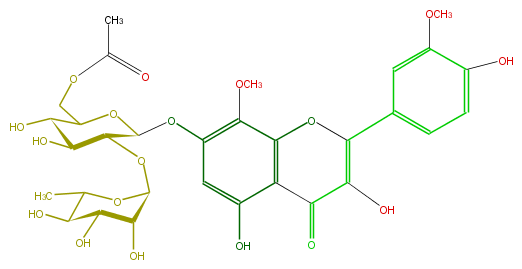
Mol:FL5FFCGS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 49 53 0 0 0 0 0 0 0 0999 V2000 | + | 49 53 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1615 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1615 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1615 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1615 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4409 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4409 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2797 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2797 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2797 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2797 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4409 -0.2979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4409 -0.2979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0002 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0002 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7208 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7208 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7208 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7208 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0002 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0002 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0002 -2.7808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0002 -2.7808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6282 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6282 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3626 -0.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3626 -0.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0970 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0970 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0970 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0970 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3626 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3626 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6282 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6282 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4409 -2.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4409 -2.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8152 0.9027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8152 0.9027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8025 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8025 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4504 -2.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4504 -2.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2284 -0.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2284 -0.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7798 -0.8590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7798 -0.8590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1340 -0.6078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1340 -0.6078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4610 -0.6152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4610 -0.6152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9637 -0.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9637 -0.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6233 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6233 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8152 -0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8152 -0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3509 -0.7059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3509 -0.7059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3856 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3856 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0363 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0363 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5363 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5363 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8734 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8734 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2250 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2250 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5727 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5727 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2356 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2356 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8840 -1.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8840 -1.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2494 -1.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2494 -1.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4321 -2.1598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4321 -2.1598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6282 -2.7342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6282 -2.7342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5577 -3.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5577 -3.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7521 0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7521 0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0721 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0721 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3252 0.5905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3252 0.5905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0721 1.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0721 1.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3626 1.8412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3626 1.8412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9343 3.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9343 3.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4409 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4409 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1246 1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1246 1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 30 1 0 0 0 0 | + | 36 30 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 31 42 1 0 0 0 0 | + | 31 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 16 46 1 0 0 0 0 | + | 16 46 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 6 48 1 0 0 0 0 | + | 6 48 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 46 47 | + | M SAL 1 2 46 47 |
| − | M SBL 1 1 51 | + | M SBL 1 1 51 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 51 0.0000 -0.7069 | + | M SBV 1 51 0.0000 -0.7069 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 48 49 | + | M SAL 2 2 48 49 |
| − | M SBL 2 1 53 | + | M SBL 2 1 53 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 53 0.0000 -0.7372 | + | M SBV 2 53 0.0000 -0.7372 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FFCGS0023 | + | ID FL5FFCGS0023 |
| − | FORMULA C31H36O18 | + | FORMULA C31H36O18 |
| − | EXACTMASS 696.190164348 | + | EXACTMASS 696.190164348 |
| − | AVERAGEMASS 696.60674 | + | AVERAGEMASS 696.60674 |
| − | SMILES C(C1O)(OC(C2Oc(c3OC)cc(c(C(=O)5)c(OC(=C5O)c(c4)cc(c(O)c4)OC)3)O)C(O)C(C(O2)COC(C)=O)O)OC(C)C(O)C(O)1 | + | SMILES C(C1O)(OC(C2Oc(c3OC)cc(c(C(=O)5)c(OC(=C5O)c(c4)cc(c(O)c4)OC)3)O)C(O)C(C(O2)COC(C)=O)O)OC(C)C(O)C(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
49 53 0 0 0 0 0 0 0 0999 V2000
-1.1615 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1615 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4409 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2797 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2797 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4409 -0.2979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0002 -1.9619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7208 -1.5459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7208 -0.7139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0002 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0002 -2.7808 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6282 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3626 -0.5617 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0970 -0.1378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0970 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3626 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6282 0.7103 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4409 -2.7938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8152 0.9027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8025 -0.2979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4504 -2.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2284 -0.2669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7798 -0.8590 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1340 -0.6078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4610 -0.6152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9637 -0.1481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6233 -0.3850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8152 -0.4241 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3509 -0.7059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3856 -1.0884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0363 -0.0094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5363 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8734 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2250 -2.1364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5727 -2.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2356 -1.7377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8840 -1.9229 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2494 -1.7910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4321 -2.1598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6282 -2.7342 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5577 -3.0035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7521 0.5360 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0721 1.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3252 0.5905 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0721 1.7189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3626 1.8412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9343 3.0035 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4409 0.4394 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1246 1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 30 1 0 0 0 0
25 20 1 0 0 0 0
31 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
46 47 1 0 0 0 0
16 46 1 0 0 0 0
48 49 1 0 0 0 0
6 48 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 46 47
M SBL 1 1 51
M SMT 1 OCH3
M SBV 1 51 0.0000 -0.7069
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 48 49
M SBL 2 1 53
M SMT 2 OCH3
M SBV 2 53 0.0000 -0.7372
S SKP 5
ID FL5FFCGS0023
FORMULA C31H36O18
EXACTMASS 696.190164348
AVERAGEMASS 696.60674
SMILES C(C1O)(OC(C2Oc(c3OC)cc(c(C(=O)5)c(OC(=C5O)c(c4)cc(c(O)c4)OC)3)O)C(O)C(C(O2)COC(C)=O)O)OC(C)C(O)C(O)1
M END