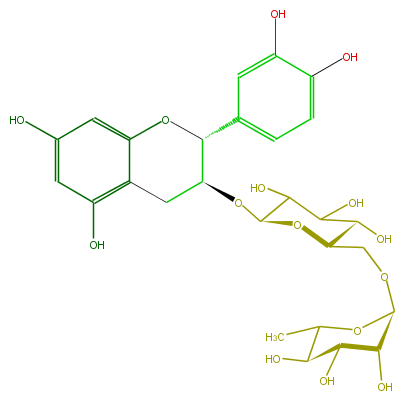
Mol:FL63ACGS0008
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0058 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0058 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0058 0.3613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0058 0.3613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2799 -0.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2799 -0.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5540 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5540 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5540 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5540 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2799 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2799 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8283 -0.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8283 -0.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1024 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1024 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1024 1.1996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1024 1.1996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8283 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8283 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6234 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6234 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3554 1.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3554 1.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0872 1.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0872 1.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0873 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0873 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3554 2.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3554 2.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6235 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6235 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7878 2.8682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7878 2.8682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7316 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7316 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6234 -0.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6234 -0.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3553 3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3553 3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2800 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2800 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2473 -2.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2473 -2.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9902 -3.2449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9902 -3.2449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6661 -2.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6661 -2.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4182 -2.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4182 -2.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7316 -2.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7316 -2.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9993 -2.6156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9993 -2.6156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4693 -2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4693 -2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6366 0.0483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6366 0.0483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0608 -0.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0608 -0.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8106 -0.4879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8106 -0.4879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4235 -0.9307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4235 -0.9307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9993 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9993 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2495 -0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2495 -0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0982 0.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0982 0.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8986 -0.0431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8986 -0.0431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1209 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1209 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5535 -1.5221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5535 -1.5221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3830 -3.1717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3830 -3.1717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3251 -3.6122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3251 -3.6122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4839 -3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4839 -3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4595 -0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4595 -0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 6 0 0 0 | + | 9 11 1 6 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 17 14 1 0 0 0 0 | + | 17 14 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 26 38 1 0 0 0 0 | + | 26 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 23 39 1 0 0 0 0 | + | 23 39 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 30 19 1 0 0 0 0 | + | 30 19 1 0 0 0 0 |
| − | 25 41 1 0 0 0 0 | + | 25 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL63ACGS0008 | + | ID FL63ACGS0008 |
| − | FORMULA C27H34O15 | + | FORMULA C27H34O15 |
| − | EXACTMASS 598.189770418 | + | EXACTMASS 598.189770418 |
| − | AVERAGEMASS 598.5498600000001 | + | AVERAGEMASS 598.5498600000001 |
| − | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2OC(C(c(c5)ccc(c(O)5)O)3)Cc(c4O)c(cc(O)c4)O3)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2OC(C(c(c5)ccc(c(O)5)O)3)Cc(c4O)c(cc(O)c4)O3)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-3.0058 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0058 0.3613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2799 -0.0577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5540 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5540 1.1995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2799 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8283 -0.0576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1024 0.3614 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1024 1.1996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8283 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6234 1.6186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3554 1.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0872 1.6187 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0873 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3554 2.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6235 2.4638 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7878 2.8682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7316 1.6186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6234 -0.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3553 3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2800 -0.8938 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2473 -2.5384 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9902 -3.2449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6661 -2.9160 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4182 -2.9931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7316 -2.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9993 -2.6156 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4693 -2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6366 0.0483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0608 -0.4347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8106 -0.4879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4235 -0.9307 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9993 -0.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2495 -0.3943 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0982 0.2590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8986 -0.0431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1209 -0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5535 -1.5221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3830 -3.1717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3251 -3.6122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4839 -3.7303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4595 -0.7607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
9 11 1 6 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
17 14 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 1 0 0 0
15 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
34 36 1 0 0 0 0
32 37 1 0 0 0 0
26 38 1 0 0 0 0
37 38 1 0 0 0 0
23 39 1 0 0 0 0
24 40 1 0 0 0 0
30 19 1 0 0 0 0
25 41 1 0 0 0 0
33 42 1 0 0 0 0
S SKP 5
ID FL63ACGS0008
FORMULA C27H34O15
EXACTMASS 598.189770418
AVERAGEMASS 598.5498600000001
SMILES O(C1C)C(OCC(O2)C(O)C(C(O)C2OC(C(c(c5)ccc(c(O)5)O)3)Cc(c4O)c(cc(O)c4)O3)O)C(C(O)C(O)1)O
M END