Mol:FL63ACNS0015
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5667 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5667 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5667 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0282 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0282 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4896 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4896 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4896 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4896 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0282 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0282 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0489 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0489 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5874 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5874 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5874 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5874 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0489 0.9657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0489 0.9657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1049 0.9656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1049 0.9656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0984 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0984 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0670 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0670 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6117 0.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6117 0.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1564 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1564 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1564 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1564 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6117 1.8752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6117 1.8752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0670 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0670 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7546 1.9200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7546 1.9200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0282 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0282 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6117 2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6117 2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6096 -1.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6096 -1.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6096 -0.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6096 -0.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2919 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2919 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5951 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5951 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2016 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2016 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5048 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5048 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2016 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2016 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5951 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5951 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5048 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5048 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1112 -0.9184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1112 -0.9184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5048 -1.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5048 -1.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5220 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5220 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1878 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1878 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2137 -1.9271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2137 -1.9271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5084 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5084 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1498 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1498 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4705 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4705 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1498 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1498 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5084 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5084 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4702 -0.2829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4702 -0.2829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1112 -1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1112 -1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4702 -2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4702 -2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 1 11 1 0 0 0 0 | + | 1 11 1 0 0 0 0 |
| − | 8 12 1 6 0 0 0 | + | 8 12 1 6 0 0 0 |
| − | 9 13 1 6 0 0 0 | + | 9 13 1 6 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 19 16 1 0 0 0 0 | + | 19 16 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 17 21 1 0 0 0 0 | + | 17 21 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 2 0 0 0 0 | + | 24 25 2 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 2 0 0 0 0 | + | 26 27 2 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 2 0 0 0 0 | + | 28 29 2 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 23 22 2 0 0 0 0 | + | 23 22 2 0 0 0 0 |
| − | 23 12 1 0 0 0 0 | + | 23 12 1 0 0 0 0 |
| − | 20 33 1 0 0 0 0 | + | 20 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 33 35 2 0 0 0 0 | + | 33 35 2 0 0 0 0 |
| − | 34 36 2 0 0 0 0 | + | 34 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 34 1 0 0 0 0 | + | 40 34 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 38 42 1 0 0 0 0 | + | 38 42 1 0 0 0 0 |
| − | 39 43 1 0 0 0 0 | + | 39 43 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL63ACNS0015 | + | ID FL63ACNS0015 |
| − | KNApSAcK_ID C00008875 | + | KNApSAcK_ID C00008875 |
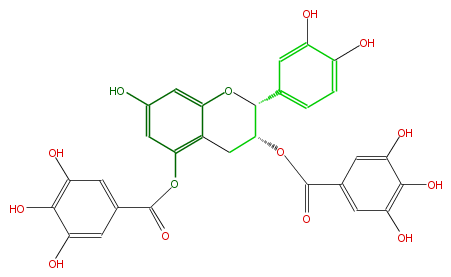
| − | NAME Epicatechin 3,5-di-O-gallate | + | NAME Epicatechin 3,5-di-O-gallate |
| − | CAS_RN 37484-74-5 | + | CAS_RN 37484-74-5 |
| − | FORMULA C29H22O14 | + | FORMULA C29H22O14 |
| − | EXACTMASS 594.100955412 | + | EXACTMASS 594.100955412 |
| − | AVERAGEMASS 594.47658 | + | AVERAGEMASS 594.47658 |
| − | SMILES c(c1)(c(c(cc1C(OC(C(c(c5)cc(c(c5)O)O)4)Cc(c3O4)c(cc(O)c3)OC(c(c2)cc(c(c2O)O)O)=O)=O)O)O)O | + | SMILES c(c1)(c(c(cc1C(OC(C(c(c5)cc(c(c5)O)O)4)Cc(c3O4)c(cc(O)c3)OC(c(c2)cc(c(c2O)O)O)=O)=O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-1.5667 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5667 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0282 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4896 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4896 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0282 0.9657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0489 -0.2779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5874 0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5874 0.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0489 0.9657 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1049 0.9656 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0984 -0.2620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0670 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6117 0.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1564 0.9317 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1564 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6117 1.8752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0670 1.5607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7546 1.9200 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0282 -0.8994 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6117 2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6096 -1.5946 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6096 -0.9333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2919 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5951 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2016 -1.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5048 -0.9184 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2016 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5951 -0.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5048 0.1320 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1112 -0.9184 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5048 -1.9687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5220 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1878 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2137 -1.9271 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5084 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1498 -0.8378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4705 -1.3932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1498 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5084 -1.9486 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4702 -0.2829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1112 -1.3932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4702 -2.5035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
1 11 1 0 0 0 0
8 12 1 6 0 0 0
9 13 1 6 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
19 16 1 0 0 0 0
3 20 1 0 0 0 0
17 21 1 0 0 0 0
23 24 1 0 0 0 0
24 25 2 0 0 0 0
25 26 1 0 0 0 0
26 27 2 0 0 0 0
27 28 1 0 0 0 0
28 29 2 0 0 0 0
29 24 1 0 0 0 0
28 30 1 0 0 0 0
27 31 1 0 0 0 0
26 32 1 0 0 0 0
23 22 2 0 0 0 0
23 12 1 0 0 0 0
20 33 1 0 0 0 0
33 34 1 0 0 0 0
33 35 2 0 0 0 0
34 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 34 1 0 0 0 0
37 41 1 0 0 0 0
38 42 1 0 0 0 0
39 43 1 0 0 0 0
S SKP 8
ID FL63ACNS0015
KNApSAcK_ID C00008875
NAME Epicatechin 3,5-di-O-gallate
CAS_RN 37484-74-5
FORMULA C29H22O14
EXACTMASS 594.100955412
AVERAGEMASS 594.47658
SMILES c(c1)(c(c(cc1C(OC(C(c(c5)cc(c(c5)O)O)4)Cc(c3O4)c(cc(O)c3)OC(c(c2)cc(c(c2O)O)O)=O)=O)O)O)O
M END