Mol:FL7AAAGL0057
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 66 72 0 0 0 0 0 0 0 0999 V2000 | + | 66 72 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6619 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6619 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6619 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6619 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9474 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9474 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2329 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2329 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2329 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2329 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9474 3.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9474 3.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4815 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4815 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1960 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1960 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1960 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1960 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4815 3.1680 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.4815 3.1680 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9725 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9725 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6870 2.7914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6870 2.7914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4015 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4015 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4015 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4015 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6870 4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6870 4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9725 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9725 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0417 4.3985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0417 4.3985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2419 3.0904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2419 3.0904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9474 0.7554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9474 0.7554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8462 1.5551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8462 1.5551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4099 0.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4099 0.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9973 0.0728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9973 0.0728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1990 0.2820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1990 0.2820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4055 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4055 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8181 0.7699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8181 0.7699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6165 0.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6165 0.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7734 1.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7734 1.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2533 1.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2533 1.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8931 0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8931 0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4460 0.3319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4460 0.3319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3704 -0.3580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3704 -0.3580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6700 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6700 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4951 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4951 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7295 0.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7295 0.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3345 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3345 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5094 0.8128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5094 0.8128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2748 0.0215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2748 0.0215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6924 0.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6924 0.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2069 -0.0765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2069 -0.0765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8332 -1.2035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8332 -1.2035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0411 -0.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0411 -0.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1882 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1882 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5599 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5599 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9855 -3.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9855 -3.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2640 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2640 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6759 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6759 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4998 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4998 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9123 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9123 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7373 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7373 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1498 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1498 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7373 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7373 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9123 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9123 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9737 -3.7269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9737 -3.7269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3155 -2.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3155 -2.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0417 -2.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0417 -2.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4899 -2.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4899 -2.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2941 -2.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2941 -2.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3279 -1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3279 -1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7773 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7773 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9730 -1.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9730 -1.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9391 -1.9972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9391 -1.9972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3329 -1.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3329 -1.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9275 -2.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9275 -2.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8103 -3.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8103 -3.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9150 -2.8574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9150 -2.8574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8896 -2.0311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8896 -2.0311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 32 40 1 0 0 0 0 | + | 32 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 20 1 0 0 0 0 | + | 35 20 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 49 54 1 0 0 0 0 | + | 49 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 65 1 0 0 0 0 | + | 57 65 1 0 0 0 0 |
| − | 58 66 1 0 0 0 0 | + | 58 66 1 0 0 0 0 |
| − | 59 42 1 0 0 0 0 | + | 59 42 1 0 0 0 0 |
| − | 43 63 1 0 0 0 0 | + | 43 63 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AAAGL0057 | + | ID FL7AAAGL0057 |
| − | KNApSAcK_ID C00014833 | + | KNApSAcK_ID C00014833 |
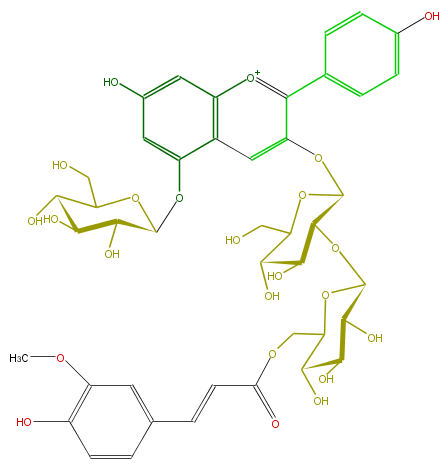
| − | NAME Pelargonidin 3-O-[2-O-(6-O-(E)-feruloyl-beta-D-glucopyranosyl)-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelaronidin 3-(2-(6-ferulylglucosyl)glucoside)-5-glucoside | + | NAME Pelargonidin 3-O-[2-O-(6-O-(E)-feruloyl-beta-D-glucopyranosyl)-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelaronidin 3-(2-(6-ferulylglucosyl)glucoside)-5-glucoside |
| − | CAS_RN 214117-66-5 | + | CAS_RN 214117-66-5 |
| − | FORMULA C43H49O23 | + | FORMULA C43H49O23 |
| − | EXACTMASS 933.266462874 | + | EXACTMASS 933.266462874 |
| − | AVERAGEMASS 933.83536 | + | AVERAGEMASS 933.83536 |
| − | SMILES C(=CC(OCC(O2)C(C(C(C2OC(C(Oc(c6)c(c(c7)ccc(O)c7)[o+1]c(c64)cc(cc4OC(C(O)5)OC(CO)C(O)C(O)5)O)3)C(C(C(CO)O3)O)O)O)O)O)=O)c(c1)ccc(O)c(OC)1 | + | SMILES C(=CC(OCC(O2)C(C(C(C2OC(C(Oc(c6)c(c(c7)ccc(O)c7)[o+1]c(c64)cc(cc4OC(C(O)5)OC(CO)C(O)C(O)5)O)3)C(C(C(CO)O3)O)O)O)O)O)=O)c(c1)ccc(O)c(OC)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
66 72 0 0 0 0 0 0 0 0999 V2000
-1.6619 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6619 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9474 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2329 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2329 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9474 3.1680 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4815 1.5180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1960 1.9305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1960 2.7555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4815 3.1680 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.9725 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6870 2.7914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4015 3.2039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4015 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6870 4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9725 4.0289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0417 4.3985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2419 3.0904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9474 0.7554 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8462 1.5551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4099 0.7875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9973 0.0728 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1990 0.2820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4055 0.0551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8181 0.7699 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6165 0.5608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7734 1.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2533 1.4237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8931 0.3043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4460 0.3319 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3704 -0.3580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6700 -0.5400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4951 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7295 0.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3345 0.7956 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5094 0.8128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2748 0.0215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6924 0.1905 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2069 -0.0765 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8332 -1.2035 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0411 -0.8067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1882 -0.2438 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5599 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9855 -3.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2640 -3.0134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6759 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4998 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9123 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7373 -3.0124 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1498 -3.7269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7373 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9123 -4.4414 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9737 -3.7269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3155 -2.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0417 -2.4343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4899 -2.6895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2941 -2.5043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3279 -1.6797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7773 -0.9875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9730 -1.1726 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9391 -1.9972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3329 -1.9757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9275 -2.3534 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8103 -3.2931 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9150 -2.8574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8896 -2.0311 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
8 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 19 1 0 0 0 0
32 33 1 1 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
35 36 1 0 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
32 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 20 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
49 54 1 0 0 0 0
54 55 1 0 0 0 0
56 57 1 1 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
59 60 1 0 0 0 0
60 61 1 0 0 0 0
61 56 1 0 0 0 0
61 62 1 0 0 0 0
62 63 1 0 0 0 0
56 64 1 0 0 0 0
57 65 1 0 0 0 0
58 66 1 0 0 0 0
59 42 1 0 0 0 0
43 63 1 0 0 0 0
S SKP 8
ID FL7AAAGL0057
KNApSAcK_ID C00014833
NAME Pelargonidin 3-O-[2-O-(6-O-(E)-feruloyl-beta-D-glucopyranosyl)-beta-D-glucopyranoside]-5-O-(beta-D-glucopyranoside);Pelaronidin 3-(2-(6-ferulylglucosyl)glucoside)-5-glucoside
CAS_RN 214117-66-5
FORMULA C43H49O23
EXACTMASS 933.266462874
AVERAGEMASS 933.83536
SMILES C(=CC(OCC(O2)C(C(C(C2OC(C(Oc(c6)c(c(c7)ccc(O)c7)[o+1]c(c64)cc(cc4OC(C(O)5)OC(CO)C(O)C(O)5)O)3)C(C(C(CO)O3)O)O)O)O)O)=O)c(c1)ccc(O)c(OC)1
M END