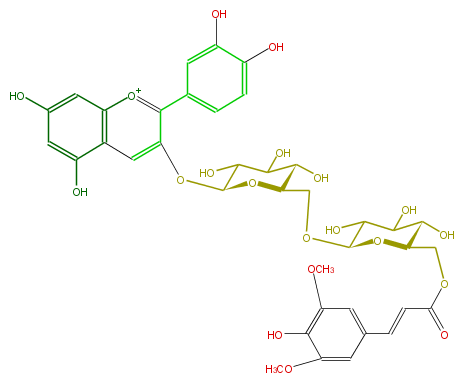
Mol:FL7AACGL0037
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 58 63 0 0 0 0 0 0 0 0999 V2000 | + | 58 63 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.4741 -0.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4741 -0.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6315 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6315 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3307 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3307 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9092 -0.7710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9092 -0.7710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4912 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4912 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7921 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7921 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2135 -0.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2135 -0.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0728 -0.5650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0728 -0.5650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4517 -0.1681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4517 -0.1681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2261 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2261 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0427 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0427 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2333 -1.6476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2333 -1.6476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9439 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9439 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4287 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4287 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1344 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1344 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6451 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6451 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3559 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3559 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7775 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7775 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4883 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4883 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7775 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7775 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3559 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3559 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9108 -2.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9108 -2.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2395 -2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2395 -2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6834 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6834 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6834 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6834 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1271 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1271 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5708 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5708 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5708 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5708 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1271 2.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1271 2.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0145 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0145 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4582 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4582 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4582 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4582 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0145 2.1238 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | -2.0145 2.1238 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9021 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9021 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3352 1.7964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3352 1.7964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2318 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2318 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2318 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2318 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3352 3.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3352 3.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9021 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9021 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2395 2.1237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2395 2.1237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7986 3.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7986 3.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1271 0.1970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1271 0.1970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0421 0.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0421 0.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1153 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1153 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1855 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1855 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3929 0.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3929 0.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9749 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9749 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2758 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2758 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6973 0.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6973 0.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4434 0.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4434 0.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9355 0.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9355 0.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7099 0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7099 0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5265 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5265 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3352 3.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3352 3.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1134 -3.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1134 -3.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8279 -3.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8279 -3.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5878 -1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5878 -1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3023 -1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3023 -1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 1 1 0 0 0 | + | 3 4 1 1 0 0 0 |
| − | 4 5 1 1 0 0 0 | + | 4 5 1 1 0 0 0 |
| − | 6 5 1 1 0 0 0 | + | 6 5 1 1 0 0 0 |
| − | 6 7 1 0 0 0 0 | + | 6 7 1 0 0 0 0 |
| − | 7 2 1 0 0 0 0 | + | 7 2 1 0 0 0 0 |
| − | 2 8 1 0 0 0 0 | + | 2 8 1 0 0 0 0 |
| − | 7 9 1 0 0 0 0 | + | 7 9 1 0 0 0 0 |
| − | 6 10 1 0 0 0 0 | + | 6 10 1 0 0 0 0 |
| − | 5 11 1 0 0 0 0 | + | 5 11 1 0 0 0 0 |
| − | 3 1 1 0 0 0 0 | + | 3 1 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 2 0 0 0 0 | + | 20 21 2 0 0 0 0 |
| − | 21 16 1 0 0 0 0 | + | 21 16 1 0 0 0 0 |
| − | 19 22 1 0 0 0 0 | + | 19 22 1 0 0 0 0 |
| − | 13 23 2 0 0 0 0 | + | 13 23 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 2 0 0 0 0 | + | 29 24 2 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 30 31 2 0 0 0 0 | + | 30 31 2 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 2 0 0 0 0 | + | 32 33 2 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 24 40 1 0 0 0 0 | + | 24 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 26 42 1 0 0 0 0 | + | 26 42 1 0 0 0 0 |
| − | 43 31 1 0 0 0 0 | + | 43 31 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 45 43 1 0 0 0 0 | + | 45 43 1 0 0 0 0 |
| − | 53 1 1 0 0 0 0 | + | 53 1 1 0 0 0 0 |
| − | 38 54 1 0 0 0 0 | + | 38 54 1 0 0 0 0 |
| − | 20 55 1 0 0 0 0 | + | 20 55 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 18 57 1 0 0 0 0 | + | 18 57 1 0 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 55 56 | + | M SAL 1 2 55 56 |
| − | M SBL 1 1 60 | + | M SBL 1 1 60 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 60 -7.0246 5.2445 | + | M SBV 1 60 -7.0246 5.2445 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 57 58 | + | M SAL 2 2 57 58 |
| − | M SBL 2 1 62 | + | M SBL 2 1 62 |
| − | M SMT 2 ^OCH3 | + | M SMT 2 ^OCH3 |
| − | M SBV 2 62 -6.5502 6.2462 | + | M SBV 2 62 -6.5502 6.2462 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0037 | + | ID FL7AACGL0037 |
| − | KNApSAcK_ID C00006807 | + | KNApSAcK_ID C00006807 |
| − | NAME Alatanin C | + | NAME Alatanin C |
| − | CAS_RN 138688-63-8 | + | CAS_RN 138688-63-8 |
| − | FORMULA C38H41O20 | + | FORMULA C38H41O20 |
| − | EXACTMASS 817.219118752 | + | EXACTMASS 817.219118752 |
| − | AVERAGEMASS 817.72014 | + | AVERAGEMASS 817.72014 |
| − | SMILES Oc(c1)c(c2)c([o+1]c(c(c6)cc(O)c(c6)O)c2OC(O3)C(O)C(C(C3COC(C(O)5)OC(C(O)C(O)5)COC(C=Cc(c4)cc(c(O)c(OC)4)OC)=O)O)O)cc1O | + | SMILES Oc(c1)c(c2)c([o+1]c(c(c6)cc(O)c(c6)O)c2OC(O3)C(O)C(C(C3COC(C(O)5)OC(C(O)C(O)5)COC(C=Cc(c4)cc(c(O)c(OC)4)OC)=O)O)O)cc1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
58 63 0 0 0 0 0 0 0 0999 V2000
1.4741 -0.7154 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6315 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3307 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9092 -0.7710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4912 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7921 -0.4153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2135 -0.5806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0728 -0.5650 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4517 -0.1681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2261 -0.6660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0427 -0.9364 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2333 -1.6476 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9439 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4287 -2.1488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1344 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6451 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3559 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7775 -2.1577 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4883 -2.6586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7775 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3559 -3.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9108 -2.6586 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2395 -2.6609 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6834 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6834 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1271 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5708 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5708 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1271 2.1238 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0145 0.8391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4582 1.1603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4582 1.8026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0145 2.1238 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
-0.9021 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3352 1.7964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2318 2.1237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2318 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3352 3.1057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9021 2.7784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2395 2.1237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7986 3.1056 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1271 0.1970 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0421 0.4396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1153 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1855 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3929 0.3840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9749 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2758 0.7397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6973 0.5744 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4434 0.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9355 0.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7099 0.4890 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5265 0.2186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3352 3.7602 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1134 -3.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8279 -3.7602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5878 -1.3442 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3023 -1.7567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2 3 1 0 0 0 0
3 4 1 1 0 0 0
4 5 1 1 0 0 0
6 5 1 1 0 0 0
6 7 1 0 0 0 0
7 2 1 0 0 0 0
2 8 1 0 0 0 0
7 9 1 0 0 0 0
6 10 1 0 0 0 0
5 11 1 0 0 0 0
3 1 1 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 20 1 0 0 0 0
20 21 2 0 0 0 0
21 16 1 0 0 0 0
19 22 1 0 0 0 0
13 23 2 0 0 0 0
24 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 24 2 0 0 0 0
27 30 1 0 0 0 0
30 31 2 0 0 0 0
31 32 1 0 0 0 0
32 33 2 0 0 0 0
33 28 1 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
24 40 1 0 0 0 0
37 41 1 0 0 0 0
26 42 1 0 0 0 0
43 31 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
44 50 1 0 0 0 0
49 51 1 0 0 0 0
48 52 1 0 0 0 0
47 53 1 0 0 0 0
45 43 1 0 0 0 0
53 1 1 0 0 0 0
38 54 1 0 0 0 0
20 55 1 0 0 0 0
55 56 1 0 0 0 0
18 57 1 0 0 0 0
57 58 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 55 56
M SBL 1 1 60
M SMT 1 ^OCH3
M SBV 1 60 -7.0246 5.2445
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 57 58
M SBL 2 1 62
M SMT 2 ^OCH3
M SBV 2 62 -6.5502 6.2462
S SKP 8
ID FL7AACGL0037
KNApSAcK_ID C00006807
NAME Alatanin C
CAS_RN 138688-63-8
FORMULA C38H41O20
EXACTMASS 817.219118752
AVERAGEMASS 817.72014
SMILES Oc(c1)c(c2)c([o+1]c(c(c6)cc(O)c(c6)O)c2OC(O3)C(O)C(C(C3COC(C(O)5)OC(C(O)C(O)5)COC(C=Cc(c4)cc(c(O)c(OC)4)OC)=O)O)O)cc1O
M END