Mol:FL7AACGL0049
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3392 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3392 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3392 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3392 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7829 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7829 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2266 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2266 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2266 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2266 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7829 0.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7829 0.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3297 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3297 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8860 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8860 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8860 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8860 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3297 0.1152 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.3297 0.1152 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4421 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4421 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0091 -0.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0091 -0.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5760 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5760 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5760 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5760 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0091 1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0091 1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4421 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4421 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8953 0.1151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8953 0.1151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1429 1.0971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1429 1.0971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7829 -1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7829 -1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3021 -1.5690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3021 -1.5690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0091 1.7517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0091 1.7517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9417 -2.0858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9417 -2.0858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5705 -2.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5705 -2.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0360 -2.3679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0360 -2.3679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5203 -2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5203 -2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8950 -1.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8950 -1.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3626 -2.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3626 -2.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4555 -2.3825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4555 -2.3825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1491 -2.6039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1491 -2.6039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7297 -2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7297 -2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8275 -1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8275 -1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5626 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5626 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2618 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2618 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8403 -1.5834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8403 -1.5834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4223 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4223 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7232 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7232 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1446 -1.3930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1446 -1.3930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0039 -1.3774 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0039 -1.3774 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3828 -0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3828 -0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4265 -1.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4265 -1.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8275 -1.0309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8275 -1.0309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4772 -0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4772 -0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0025 -0.9424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0025 -0.9424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4700 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4700 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9342 0.3866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9342 0.3866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9342 0.9214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9342 0.9214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3681 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3681 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3681 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3681 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9342 2.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9342 2.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5004 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5004 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5004 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5004 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9342 2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9342 2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7859 -1.7618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7859 -1.7618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5004 -2.1743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5004 -2.1743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 8 1 0 0 0 0 | + | 20 8 1 0 0 0 0 |
| − | 15 21 1 0 0 0 0 | + | 15 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 27 31 1 0 0 0 0 | + | 27 31 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 20 1 0 0 0 0 | + | 33 20 1 0 0 0 0 |
| − | 31 41 1 0 0 0 0 | + | 31 41 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 41 1 0 0 0 0 | + | 42 41 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 35 53 1 0 0 0 0 | + | 35 53 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 58 -5.1377 3.4522 | + | M SBV 1 58 -5.1377 3.4522 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0049 | + | ID FL7AACGL0049 |
| − | KNApSAcK_ID C00006820 | + | KNApSAcK_ID C00006820 |
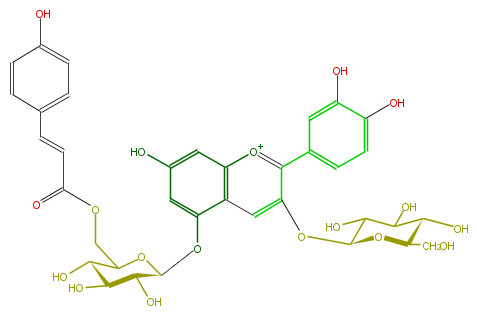
| − | NAME Gentiocyanin C;Cyanidin 3-glucoside-5-(6-p-coumaroylglucoside) | + | NAME Gentiocyanin C;Cyanidin 3-glucoside-5-(6-p-coumaroylglucoside) |
| − | CAS_RN 170900-28-4 | + | CAS_RN 170900-28-4 |
| − | FORMULA C36H37O18 | + | FORMULA C36H37O18 |
| − | EXACTMASS 757.1979893800001 | + | EXACTMASS 757.1979893800001 |
| − | AVERAGEMASS 757.66818 | + | AVERAGEMASS 757.66818 |
| − | SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O | + | SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.3392 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3392 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7829 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2266 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2266 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7829 0.1152 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3297 -1.1695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8860 -0.8483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8860 -0.2059 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3297 0.1152 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.4421 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0091 -0.2122 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5760 0.1151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5760 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0091 1.0972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4421 0.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8953 0.1151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1429 1.0971 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7829 -1.8116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3021 -1.5690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0091 1.7517 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9417 -2.0858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5705 -2.5758 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0360 -2.3679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5203 -2.3623 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8950 -1.9875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3626 -2.2343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4555 -2.3825 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1491 -2.6039 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7297 -2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8275 -1.7322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5626 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2618 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8403 -1.5834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4223 -1.7487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7232 -1.2277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1446 -1.3930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0039 -1.3774 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3828 -0.9804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4265 -1.4161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8275 -1.0309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4772 -0.6391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0025 -0.9424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4700 0.1185 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9342 0.3866 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9342 0.9214 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3681 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3681 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9342 2.2289 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5004 1.9020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5004 1.2483 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9342 2.8820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7859 -1.7618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5004 -2.1743 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 8 1 0 0 0 0
15 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
27 31 1 0 0 0 0
25 19 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 20 1 0 0 0 0
31 41 1 0 0 0 0
42 43 2 0 0 0 0
42 41 1 0 0 0 0
42 44 1 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 46 1 0 0 0 0
49 52 1 0 0 0 0
35 53 1 0 0 0 0
53 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 58
M SMT 1 CH2OH
M SBV 1 58 -5.1377 3.4522
S SKP 8
ID FL7AACGL0049
KNApSAcK_ID C00006820
NAME Gentiocyanin C;Cyanidin 3-glucoside-5-(6-p-coumaroylglucoside)
CAS_RN 170900-28-4
FORMULA C36H37O18
EXACTMASS 757.1979893800001
AVERAGEMASS 757.66818
SMILES C(C(C6O)OC(C(C(O)6)O)Oc(c2)c(c4)c([o+1]c(c(OC(O5)C(C(O)C(C(CO)5)O)O)4)c(c3)ccc(O)c(O)3)cc2O)OC(C=Cc(c1)ccc(c1)O)=O
M END