Mol:FL7AACGL0111
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 77 84 0 0 0 0 0 0 0 0999 V2000 | + | 77 84 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5074 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5074 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5074 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5074 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7929 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7929 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0784 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0784 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0784 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0784 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7929 3.3903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7929 3.3903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6360 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6360 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3505 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3505 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3505 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3505 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6360 3.3903 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.6360 3.3903 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1271 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1271 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8415 3.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8415 3.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5560 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5560 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5560 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5560 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8415 4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8415 4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1271 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1271 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1962 4.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1962 4.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9947 1.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9947 1.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0873 3.3126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0873 3.3126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7929 0.9776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7929 0.9776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2009 3.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2009 3.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3049 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3049 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8923 0.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8923 0.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0939 0.5699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0939 0.5699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3005 0.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3005 0.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7130 1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7130 1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5114 0.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5114 0.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6684 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6684 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1483 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1483 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7881 0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7881 0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3410 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3410 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2654 -0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2654 -0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3967 -0.8228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3967 -0.8228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1911 -0.5992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1911 -0.5992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1853 0.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1853 0.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6009 0.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6009 0.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8065 0.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8065 0.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8121 -0.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8121 -0.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2057 -0.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2057 -0.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7457 -1.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7457 -1.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8294 -0.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8294 -0.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7632 -0.0980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7632 -0.0980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7643 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7643 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3327 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3327 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7657 -2.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7657 -2.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4912 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4912 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9031 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9031 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7270 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7270 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1395 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1395 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9645 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9645 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3770 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3770 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9645 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9645 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1395 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1395 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2009 -2.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2009 -2.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3765 -0.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3765 -0.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6205 -2.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6205 -2.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4137 -2.5113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4137 -2.5113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4038 -1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4038 -1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8158 -0.9710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8158 -0.9710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0225 -1.1985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0225 -1.1985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0323 -2.0237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0323 -2.0237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4259 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4259 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0411 -2.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0411 -2.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9725 -3.3246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9725 -3.3246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0539 -2.8840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0539 -2.8840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9833 -2.0072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9833 -2.0072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6211 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6211 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0400 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0400 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7972 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7972 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3853 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3853 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4386 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4386 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8511 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8511 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6761 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6761 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0886 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0886 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6761 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6761 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8511 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8511 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9125 -3.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9125 -3.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 14 17 1 0 0 0 0 | + | 14 17 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 13 21 1 0 0 0 0 | + | 13 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 18 1 0 0 0 0 | + | 36 18 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 44 46 1 0 0 0 0 | + | 44 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 48 1 0 0 0 0 | + | 53 48 1 0 0 0 0 |
| − | 51 54 1 0 0 0 0 | + | 51 54 1 0 0 0 0 |
| − | 50 55 1 0 0 0 0 | + | 50 55 1 0 0 0 0 |
| − | 43 39 1 0 0 0 0 | + | 43 39 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 61 56 1 0 0 0 0 | + | 61 56 1 0 0 0 0 |
| − | 61 62 1 0 0 0 0 | + | 61 62 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 56 64 1 0 0 0 0 | + | 56 64 1 0 0 0 0 |
| − | 57 65 1 0 0 0 0 | + | 57 65 1 0 0 0 0 |
| − | 58 66 1 0 0 0 0 | + | 58 66 1 0 0 0 0 |
| − | 59 42 1 0 0 0 0 | + | 59 42 1 0 0 0 0 |
| − | 67 68 2 0 0 0 0 | + | 67 68 2 0 0 0 0 |
| − | 67 69 1 0 0 0 0 | + | 67 69 1 0 0 0 0 |
| − | 69 70 2 0 0 0 0 | + | 69 70 2 0 0 0 0 |
| − | 70 71 1 0 0 0 0 | + | 70 71 1 0 0 0 0 |
| − | 71 72 2 0 0 0 0 | + | 71 72 2 0 0 0 0 |
| − | 72 73 1 0 0 0 0 | + | 72 73 1 0 0 0 0 |
| − | 73 74 2 0 0 0 0 | + | 73 74 2 0 0 0 0 |
| − | 74 75 1 0 0 0 0 | + | 74 75 1 0 0 0 0 |
| − | 75 76 2 0 0 0 0 | + | 75 76 2 0 0 0 0 |
| − | 76 71 1 0 0 0 0 | + | 76 71 1 0 0 0 0 |
| − | 74 77 1 0 0 0 0 | + | 74 77 1 0 0 0 0 |
| − | 63 67 1 0 0 0 0 | + | 63 67 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL7AACGL0111 | + | ID FL7AACGL0111 |
| − | KNApSAcK_ID C00014771 | + | KNApSAcK_ID C00014771 |
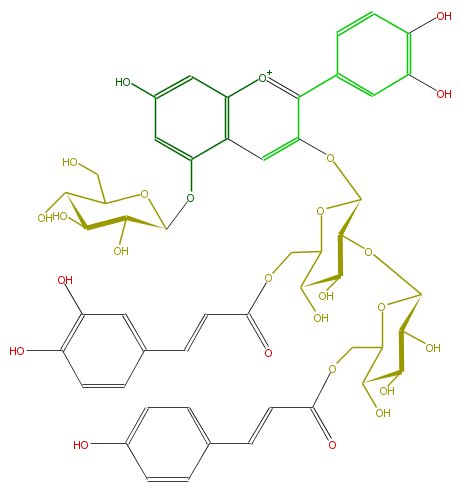
| − | NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-caffeoylglucoside]-5-glucoside | + | NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-caffeoylglucoside]-5-glucoside |
| − | CAS_RN 611232-15-6 | + | CAS_RN 611232-15-6 |
| − | FORMULA C51H53O26 | + | FORMULA C51H53O26 |
| − | EXACTMASS 1081.282506868 | + | EXACTMASS 1081.282506868 |
| − | AVERAGEMASS 1081.95092 | + | AVERAGEMASS 1081.95092 |
| − | SMILES O(C(O8)C(C(O)C(C8CO)O)O)c(c71)cc(O)cc([o+1]c(c(c7)OC(O3)C(OC(O5)C(O)C(O)C(O)C5COC(=O)C=Cc(c6)ccc(c6)O)C(C(O)C3COC(=O)C=Cc(c4)cc(O)c(c4)O)O)c(c2)ccc(O)c2O)1 | + | SMILES O(C(O8)C(C(O)C(C8CO)O)O)c(c71)cc(O)cc([o+1]c(c(c7)OC(O3)C(OC(O5)C(O)C(O)C(O)C5COC(=O)C=Cc(c6)ccc(c6)O)C(C(O)C3COC(=O)C=Cc(c4)cc(O)c(c4)O)O)c(c2)ccc(O)c2O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
77 84 0 0 0 0 0 0 0 0999 V2000
-1.5074 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5074 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7929 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0784 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0784 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7929 3.3903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6360 1.7403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3505 2.1528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3505 2.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6360 3.3903 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
2.1271 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8415 3.0136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5560 3.4261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5560 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8415 4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1271 4.2511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1962 4.6207 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9947 1.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0873 3.3126 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7929 0.9776 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2009 3.0538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3049 1.0754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8923 0.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0939 0.5699 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3005 0.3430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7130 1.0578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5114 0.8487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6684 1.4346 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1483 1.7116 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7881 0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3410 0.6198 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2654 -0.0701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3967 -0.8228 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1911 -0.5992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1853 0.2260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6009 0.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8065 0.7155 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8121 -0.1098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2057 -0.1174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7457 -1.4103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8294 -0.9701 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7632 -0.0980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7643 -0.6250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3327 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7657 -2.1225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4912 -1.3725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9031 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7270 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1395 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9645 -1.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3770 -2.0860 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9645 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1395 -2.8005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2009 -2.0860 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3765 -0.6580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6205 -2.7388 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4137 -2.5113 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4038 -1.6861 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8158 -0.9710 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0225 -1.1985 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0323 -2.0237 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4259 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0411 -2.4330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9725 -3.3246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0539 -2.8840 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9833 -2.0072 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6211 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0400 -3.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7972 -3.2356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3853 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4386 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8511 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6761 -3.2347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0886 -3.9491 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6761 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8511 -4.6636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9125 -3.9491 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
14 17 1 0 0 0 0
8 18 1 0 0 0 0
1 19 1 0 0 0 0
3 20 1 0 0 0 0
13 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 20 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
35 42 1 0 0 0 0
36 18 1 0 0 0 0
43 44 1 0 0 0 0
44 45 2 0 0 0 0
44 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 48 1 0 0 0 0
51 54 1 0 0 0 0
50 55 1 0 0 0 0
43 39 1 0 0 0 0
56 57 1 1 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
59 60 1 0 0 0 0
60 61 1 0 0 0 0
61 56 1 0 0 0 0
61 62 1 0 0 0 0
62 63 1 0 0 0 0
56 64 1 0 0 0 0
57 65 1 0 0 0 0
58 66 1 0 0 0 0
59 42 1 0 0 0 0
67 68 2 0 0 0 0
67 69 1 0 0 0 0
69 70 2 0 0 0 0
70 71 1 0 0 0 0
71 72 2 0 0 0 0
72 73 1 0 0 0 0
73 74 2 0 0 0 0
74 75 1 0 0 0 0
75 76 2 0 0 0 0
76 71 1 0 0 0 0
74 77 1 0 0 0 0
63 67 1 0 0 0 0
S SKP 8
ID FL7AACGL0111
KNApSAcK_ID C00014771
NAME Cyanidin 3-[2-(6-p-coumarylglucosyl)-6-caffeoylglucoside]-5-glucoside
CAS_RN 611232-15-6
FORMULA C51H53O26
EXACTMASS 1081.282506868
AVERAGEMASS 1081.95092
SMILES O(C(O8)C(C(O)C(C8CO)O)O)c(c71)cc(O)cc([o+1]c(c(c7)OC(O3)C(OC(O5)C(O)C(O)C(O)C5COC(=O)C=Cc(c6)ccc(c6)O)C(C(O)C3COC(=O)C=Cc(c4)cc(O)c(c4)O)O)c(c2)ccc(O)c2O)1
M END