Mol:FL7AADGL0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.9993 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9993 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9993 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9993 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2844 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2844 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5696 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5696 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5696 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5696 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2844 0.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2844 0.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1452 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1452 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8601 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8601 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8601 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8601 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1452 0.5979 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 | + | 0.1452 0.5979 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5747 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5747 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3032 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3032 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0318 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0318 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0318 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0318 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3032 1.8596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3032 1.8596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5747 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5747 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7138 0.5976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7138 0.5976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6548 1.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6548 1.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2844 -1.8781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2844 -1.8781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6204 -2.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6204 -2.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1754 -2.6672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1754 -2.6672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5348 -2.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5348 -2.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8670 -2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8670 -2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3658 -1.9621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3658 -1.9621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0203 -2.1970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0203 -2.1970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3432 -2.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3432 -2.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8621 -2.7265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8621 -2.7265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8556 -2.9339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8556 -2.9339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6270 -1.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6270 -1.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5688 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5688 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2241 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2241 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8870 -2.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8870 -2.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5538 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5538 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8985 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8985 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2356 -2.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2356 -2.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9066 -2.4970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9066 -2.4970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6453 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6453 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3006 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3006 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9635 -1.0763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9635 -1.0763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6303 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6303 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9750 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9750 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3121 -0.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3121 -0.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9721 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9721 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8562 -0.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8562 -0.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6726 -0.8590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6726 -0.8590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2773 -1.2052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2773 -1.2052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8296 -1.7191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8296 -1.7191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6354 -2.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6354 -2.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5239 -3.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5239 -3.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5538 -3.6032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5538 -3.6032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6716 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6716 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8985 -1.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8985 -1.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3032 2.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3032 2.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8704 3.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8704 3.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 9 11 1 0 0 0 0 | + | 9 11 1 0 0 0 0 |
| − | 11 12 2 0 0 0 0 | + | 11 12 2 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 11 1 0 0 0 0 | + | 16 11 1 0 0 0 0 |
| − | 1 17 1 0 0 0 0 | + | 1 17 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 29 8 1 0 0 0 0 | + | 29 8 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 41 40 1 1 0 0 0 | + | 41 40 1 1 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 41 45 1 0 0 0 0 | + | 41 45 1 0 0 0 0 |
| − | 40 46 1 0 0 0 0 | + | 40 46 1 0 0 0 0 |
| − | 34 47 1 0 0 0 0 | + | 34 47 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 31 48 1 0 0 0 0 | + | 31 48 1 0 0 0 0 |
| − | 32 49 1 0 0 0 0 | + | 32 49 1 0 0 0 0 |
| − | 33 50 1 0 0 0 0 | + | 33 50 1 0 0 0 0 |
| − | 38 29 1 0 0 0 0 | + | 38 29 1 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 25 51 1 0 0 0 0 | + | 25 51 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 15 53 1 0 0 0 0 | + | 15 53 1 0 0 0 0 |
| − | M CHG 1 10 1 | + | M CHG 1 10 1 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 51 52 | + | M SAL 1 2 51 52 |
| − | M SBL 1 1 57 | + | M SBL 1 1 57 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 57 0.6514 -0.5569 | + | M SBV 1 57 0.6514 -0.5569 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 53 54 | + | M SAL 2 2 53 54 |
| − | M SBL 2 1 59 | + | M SBL 2 1 59 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 59 0.0000 -0.5906 | + | M SBV 2 59 0.0000 -0.5906 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL7AADGL0010 | + | ID FL7AADGL0010 |
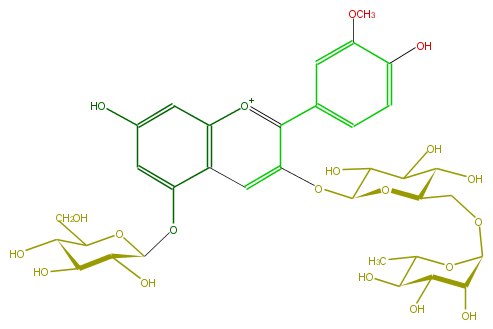
| − | FORMULA C34H43O20 | + | FORMULA C34H43O20 |
| − | EXACTMASS 771.234768816 | + | EXACTMASS 771.234768816 |
| − | AVERAGEMASS 771.6932199999999 | + | AVERAGEMASS 771.6932199999999 |
| − | SMILES O(C(C(O)6)OC(CO)C(O)C6O)c(c1)c(c3)c([o+1]c(c(OC(O4)C(O)C(O)C(O)C4COC(O5)C(C(C(C5C)O)O)O)3)c(c2)ccc(c(OC)2)O)cc(O)1 | + | SMILES O(C(C(O)6)OC(CO)C(O)C6O)c(c1)c(c3)c([o+1]c(c(OC(O4)C(O)C(O)C(O)C4COC(O5)C(C(C(C5C)O)O)O)3)c(c2)ccc(c(OC)2)O)cc(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-1.9993 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9993 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2844 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5696 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5696 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2844 0.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1452 -1.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8601 -0.6403 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8601 0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1452 0.5979 0.0000 O 0 3 0 0 0 0 0 0 0 0 0 0
1.5747 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3032 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0318 0.5976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0318 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3032 1.8596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5747 1.4389 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7138 0.5976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6548 1.7986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2844 -1.8781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6204 -2.0799 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1754 -2.6672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5348 -2.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8670 -2.4255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3658 -1.9621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0203 -2.1970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3432 -2.3510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8621 -2.7265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8556 -2.9339 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6270 -1.0572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5688 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2241 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8870 -2.8321 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5538 -3.0217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8985 -2.4247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2356 -2.6141 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9066 -2.4970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6453 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3006 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9635 -1.0763 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6303 -1.2657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9750 -0.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3121 -0.8581 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9721 -0.6966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8562 -0.2540 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6726 -0.8590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2773 -1.2052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8296 -1.7191 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6354 -2.8259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5239 -3.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5538 -3.6032 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6716 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8985 -1.2582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3032 2.4502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8704 3.6032 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 5 1 0 0 0 0
9 11 1 0 0 0 0
11 12 2 0 0 0 0
12 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 11 1 0 0 0 0
1 17 1 0 0 0 0
14 18 1 0 0 0 0
3 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 19 1 0 0 0 0
29 8 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 1 0 0 0
39 40 1 1 0 0 0
41 40 1 1 0 0 0
41 42 1 0 0 0 0
42 37 1 0 0 0 0
37 43 1 0 0 0 0
42 44 1 0 0 0 0
41 45 1 0 0 0 0
40 46 1 0 0 0 0
34 47 1 0 0 0 0
46 47 1 0 0 0 0
31 48 1 0 0 0 0
32 49 1 0 0 0 0
33 50 1 0 0 0 0
38 29 1 0 0 0 0
51 52 1 0 0 0 0
25 51 1 0 0 0 0
53 54 1 0 0 0 0
15 53 1 0 0 0 0
M CHG 1 10 1
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 51 52
M SBL 1 1 57
M SMT 1 ^ CH2OH
M SBV 1 57 0.6514 -0.5569
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 53 54
M SBL 2 1 59
M SMT 2 OCH3
M SBV 2 59 0.0000 -0.5906
S SKP 5
ID FL7AADGL0010
FORMULA C34H43O20
EXACTMASS 771.234768816
AVERAGEMASS 771.6932199999999
SMILES O(C(C(O)6)OC(CO)C(O)C6O)c(c1)c(c3)c([o+1]c(c(OC(O4)C(O)C(O)C(O)C4COC(O5)C(C(C(C5C)O)O)O)3)c(c2)ccc(c(OC)2)O)cc(O)1
M END